- Protéasome
-
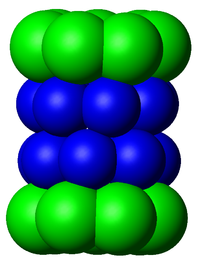
 Représentation du protéasome obtenue par Diffractométrie de rayons X après cristalisation. Le cœur catalytique 20S est en bleu. Les deux complexes régulateurs 11S sont en rouge.
Représentation du protéasome obtenue par Diffractométrie de rayons X après cristalisation. Le cœur catalytique 20S est en bleu. Les deux complexes régulateurs 11S sont en rouge.
Les protéasomes sont des complexes enzymatiques multiprotéiques que l'on retrouve chez les eucaryotes, les archées ainsi que chez quelques bactéries de l'ordre Actinomycetales. Dans les cellules eucaryotes, ils se trouvent dans le noyau, le cytosol et associés au réticulum endoplasmique[1]. Leur fonction principale est de dégrader les protéines mal repliées, dénaturées ou obsolètes de manière ciblée. Cette dégradation se fait par protéolyse, une réaction chimique qui coupe les liaisons peptiques et qui est effectuée par des enzymes appelées protéases. La protéine est ainsi découpée en peptides long de 7 à 9 acides aminés qui seront ensuite hydrolysés hors du protéasome et recyclés[2]. Les protéines sont marquées pour la dégradation par une protéine appelée ubiquitine. Ce marquage est réalisé par l'action coordonnée de trois types d'enzymes. Une fois le marquage par une première molécule d'ubiquitine réalisé, d'autres ubiquitines vont être rajoutées à sa suite. Il faudra une chaîne d'au moins quatre ubiquitines pour que le protéasome 26S reconnaisse la protéine à dégrader[2].
Le protéasome a une forme de baril et possède une cavité en son centre cernée par quatre anneaux, fournissant ainsi un espace clos pour la digestion des protéines. Chaque anneau est composé de sept protéines: les deux anneaux intérieurs sont constitués de sept sous unités β qui contiennent le site actif de la protéase, tandis que les deux anneaux extérieurs contiennent sept sous-unités α dont le rôle consiste à maintenir l'ouverture par laquelle les protéines à dégrader pénètrent dans le baril: ces sous-unités α sont capables de reconnaître les marqueurs de polyubiquitine qui régulent le processus de dégradation. L'ensemble est connu sous le terme de complexe protéasome-ubiquitine.
La dégradation protéasomale est un élément essentiel de nombreux processus cellulaires, notamment le cycle cellulaire, l'expression génétique et la réponse au stress oxydatif. L'importance de la dégradation protéolytique et du rôle de l'ubiquitine lors de celle-ci a été officialisée par la remise du Prix Nobel de chimie 2004 à Aaron Ciechanover, Avram Hershko et Irwin Rose[3]. La régulation du protéasome fait encore de nos jours l'objet de recherches intensives.
Sommaire
Découverte
Jusqu'à la découverte du complexe ubiquitine-protéasome, la dégradation des protéines dans les cellules était considérée comme essentiellement basée autour des lysosomes, des organites liés à la membrane cellulaire et dont l'intérieur acide est rempli de protéases qui peuvent dégrader et recycler les protéines exogènes et les organites usés ou endommagés[2]. De nouveaux travaux sur la dégradation protéique ATP-dépendante dans les réticulocytes, dépouvus de lysosomes, suggéraient la présence d'un mécanisme intracellulaire alternatif pour gérer cette dégradation: celui-ci, composé de plusieurs chaînes protéiques distinctes, fut décrit pour la première fois en 1978[4]. Des recherches subséquentes sur la modification des histones permit l'identification d'une modification covalente inattendue entre une lysine et une glycine N-terminale de l'ubiquitine, dont on ignorait à l'époque la fonction. On fit alors le lien avec une protéine récemment découverte et connue sous le nom de facteur protéolytique ATP-dépendant 1 (ATP-dependent proteolysis factor 1, ou plus simplement APF-1), qui ne faisait dès lors plus qu'un avec l'ubiquitine[5].
Les lauréats du Prix Nobel 2004, avec de gauche à droite: Irvin Rose, Avram Hershko et Aaron Ciechanover.
L'essentiel des premiers travaux qui menèrent à l'identification du protéasome eurent lieu dans les années 1970 et 80 au Technion, dans le laboratoire d'Avram Hershko où Aaron Ciechanover était doctorant. L'année sabbatique prise par Hershko au sein du groupe d'Irwin Rose au Fox Chase Cancer Center de Philadelphie permit de développer de nouvelles approches conceptuelles, bien que Rose ait largement relativisé son rôle dans cette découverte[6]. Quoi qu'il en soit, les trois chercheurs se sont partagés le Prix Nobel de chimie en 2004 pour leur découverte[3].
L'utilisation de la microscopie électronique au milieu des années 1980 permit de révélé la structure en anneaux empilés qui est celle du protéasome[7], mais la structure d'une des sous-unités (la 20S) ne fut résolue par cristallographie qu'en 1994[8]. Aucune structure complète montrant l'interaction entre centre actif et protéine cible n'a encore été résolue à ce jour (mars 2007), même si des modèles ont été développés qui donnent une bonne idée du fonctionnement du complexe[9].
Structure et organisation
Les composants du protéasome sont nommés d'après leur coefficient de sédimentation de Svedberg (noté S). La forme la plus répandue est le protéasome 26S de masse moléculaire 2000 kilodaltons et qui contient un cœur catalytique en forme de baril, le 20S, et deux complexes régulateurs 19S placés de chaque côté de celui-ci.
Le centre est creux et forme la cavité au cœur de laquelle les protéines sont dégradées, après être entrées par l'une des deux extrémités, celle-ci s'associant avec une sous-unité régulatrice 19S contenant des site actif d'ATPase et des sites de liaison avec l'ubiquitine. Cette structure reconnaît les protéines polyubiquitinées et les transfert au centre catalytique. Une autre sous-unité (11S) peut également s'associer avec celui-ci, de la même manière que le ferait un complexe 19S: il a été postulé que 11S jouerait alors un rôle dans la dégradation de peptides "étrangers", tels que ceux produits lors de l'infection par un virus[10].
Cœur catalytique 20S
Le nombre et la variété des sous-unités constituant le complexe 20S dépend de l'organisme dont il fait partie, étant entendu que le nombre de sous-unités distinctes et leur degré de spécialisation est plus grand chez les multicellulaires que chez les unicellulaires, ainsi que chez les eucaryotes par rapport au procaryotes. Cependant, tous les 20S sont constitués de quatre structures heptamériques circulaires, elles-mêmes composée de deux types distincts de sous-unités: les deux anneaux extérieurs de sous unités α servent pour la régulation et ont un rôle plutôt structural (régulation de l'accès des protéines au cœur du protéasome), tandis que les sept sous-unités β des deux anneaux intérieurs portent l’activité catalytique par le biais des sites actifs des protéases qu'elles contiennent.
La taille du protéasome s'est relativement bien conservée pendant l'évolution, celui-ci mesurant environ 150 angstroms (Å) de long par 115 Å de diamètre. Le centre actif ne fait pas plus de 53 Å, mais il arrive que son chemin d'accès ne dépasse pas les 13 Å de large - il est donc probable que les protéines doivent être déjà partiellement dénaturées pour pouvoir interagir avec les sites actifs[11].
Chez les eucaryotes (comme la levure), les sept sous-unités sont toutes différentes les unes des autres, tandis que chez les archées elles sont toutes semblables. Les bactéries, à l'exception de l'ordre Actinomycetales, n’ont pas de protéasome 20S mais un complexe analogue appelé Hs1v avec un site catalytique ouvert. Chez les eucaryotes, seules trois sous-unités β sont actives mais elles ont des spécificités de coupure différentes : de type trypsine pour les acides aminés basiques, chymotrypsine pour les acides aminés aromatiques et caspase pour les acides aminés acides. Chez les mammifères par exemple, ce sont les sous-unités β1, β2, et β5 qui sont catalytiques[12]. Des variantes notées β1i, β2i et β5i peuvent être exprimées dans les cellules hematopoïétiques en réponse à des signaux inflammatoires tels que les cytokines, notamment les interférons gammas. Un protéasome constitué de ces sous-unités est appelé un immunoprotéasome, dont la spécificité de substrat est altérée par rapport à un protéasome "normal"[11].
Complexe régulateur 19S
Le complexe 19S est constitué de 19 protéines distinctes et comporte deux zones : une « base » de 10 protéines se colle à l'anneau α du 20S, et possède l’activité ATPase sur 6 de ses 10 constituants (c'est la présence d'ATP qui permet l'association de 19S et 20S); l’autre partie (9 protéines), forme un « chapeau » plus flexible qui sert de couvercle à l’entrée dans le site catalytique. C’est ce complexe qui reconnaît la chaîne de polyubiquitine[13]. Il participe au dépliement de la protéine ainsi qu'à sa translocation dans le cœur du 20S, et contient également une isopeptidase qui va enlever la chaîne d’ubiquitine de la protéine, bien qu'on ne sache pas ecnore exactement si l'énergie dégagée par l'hydrolyse de l'ATP sert au dépliement[14], à l'ouverture du canal central[15] ou aux deux[13].
Les différents constituants du complexe 19S ont leur propre activité de régulation: La gankyrine, une oncoprotéine récemment découverte, est un élément de 19S qui interagit également avec la kinase cycline-dépendanteCDK4, et joue un rôle dans la reconnaissance de la version ubiquitinée de p53 par le biais de son affinité avec l'ubiquitine ligase MDM2. La gankyrine est anti-apoptotique et il a été démontré qu'elle était surexprimée dans certains types de tumeurs tels que le carcinome hépatocellulaire[16].
Complexe régulateur 11S
Il existe enfin un complexe 11S heptamérique dit « activateur » qui peut s’associer au 20S. Il stimule l’activité peptidase du protéasome mais est incapable de reconnaître l’ubiquitine et ne possède pas d'activié ATPase. Il pourrait jouer un rôle dans la dégradation des peptides viraux, mais pas de protéines plus larges. Cette structure 11S peut parfois être rencontrée sous le terme de PA28 ou REG. Le mécanisme utilisé pour se lier à 20S par le biais de son C-terminus et ouvrir l'accès au cœur du protéasome en favorisant une modification de la conformation des anneaux α semble très proche, sinon similaire, à celui utilisé par le complexe 19S[17].
L'expression de 11S est induite par l'interféron gamma et, en association avec les sous-unités β de l'immunoprotéasome, provoque la génération de peptides qui iront se lier au complexe majeur d'histocompatibilité[10].
La structure du complexe 26S n'était toujours pas élucidée en 2006, mais on pense que les complexes 11S et 19S se lient de manière similaire au complexe 20S[15].
Assemblage
L'assemblage du protéasome est un processus complexe de par le nombre de sous-unités qui doivent s'associer afin de former un complexe actif. Les sous-unités β sont synthétisées avec un "propeptide" N-terminal qui sera modifié pendant l'assemblage du complexe 20S afin d'exposer le site actif protélytique. 20S est alors assemblé à partir de deux demi-protéasomes, chacun constitué d'un proto-anneau β à sept constituants et attaché à un heptamère α.
L'association des deux anneaux β déclenche une autolyse thréonine-dépendante des propeptides, exposant ainsi le site actif. Ces interactions entre β sont essentiellement le fait de ponts salins et d'interactions hydrophobes entre des hélices alpha conservées (leur mutation diminue la faculté d'assemblage du protéasome[18]). L'association des demi-protéasomes est initiée par le regroupement des sous-unités α en heptamère. Le mode d'assemblage de ces dernières n'a pas encore été caractérisé[19].
On connaît en général beaucoup moins le mode d'assemblage et de maturation du complexe régulateur 19S. Les sous-unités pourraient s'assembler en deux composants distincts, une base d'ATPases et un chapeau ubiquitine-spécifique. Les six ATPases s'assembleraient en paires par le biais d'interactions coiled coil[20]. L'ordre dans lequel les 19 sous-unités du complexe régulateur s'assemblent est probablement lié à un mécanisme empêchant l'exposition du site actif tant que le mécanisme de régulation n'est lui-même pas en place[15].
Processus de dégradation protéique
Ubiquitination
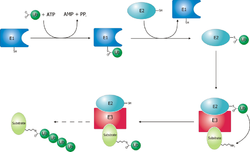
L’ubiquitine est une protéine de 76 acides aminés hautement conservée chez les Eucaryotes. Elle sert de signal de reconnaissance pour la dégradation par le protéasome 26S. Elle est fixée à la protéine à détruire par une liaison covalente entre l’un des résidus glycine de sa partie C-terminale et un groupement NH2 d’une lysine de la protéine ciblée. D’autres ubiquitines peuvent alors se fixer à un résidu lysine de la première ubiquitine pour former une chaîne. En effet, seules les protéines liées à une chaîne d’au moins quatre molécules d’ubiquitine sont reconnues par le protéasome 26S.
Ces opérations sont réalisés avec l’aide de trois autres types d’enzymes (notées E1,E2 et E3):
- L’enzyme d’activation de l’ubiquitine (E1), active l’ubiquitine en présence d’ATP en formant une liaison thiolester entre un résidu cystéine de son site catalytique et l’ubiquitine, puis transfère l’ubiquitine activée sur l’une des E2s.
- L’enzyme de conjugaison de l’ubiquitine(E2), modifie une fois de plus l’ubiquitine en formant une liaison thiolester et est capable d’attacher l’ubiquitine à la protéine cible en général avec l’aide d’une E3.
- L’ubiquitine ligase (E3), joue un rôle dans la reconnaissance entre l’ubiquitine et les différentes protéines cibles.
Un des domaines de l’ubiquitine reconnaît alors le complexe 19S du protéasome. Après avoir été enlevée par le protéasome, la chaîne de polyubiquitine est découpée pour être réutilisée. La monoubiquitination des protéines est également impliquée dans d’autres processus dont l’endocytose et l’exocytose.
Notes et références
- Peters JM, Franke WW, Kleinschmidt JA. (1994) Distinct 19S and 20S subcomplexes of the 26S proteasome and their distribution in the nucleus and the cytoplasm. J Biol Chem, Mar 11;269(10):7709-18. PMID 8125997
- Lodish, H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Cell Biology, 5th ed., ch.3, pp 66-72. New York: WH Freeman. (ISBN 0716743663)
- Gagnants du Prix Nobel 2004, sur le site du Comité Nobel
- Ciehanover A, Hod Y, Hershko A. (1978). A heat-stable polypeptide component of an ATP-dependent proteolytic system from reticulocytes. Biochemical and Biophysical Research Communications 81(4):1100-1105. PMID 666810
- Ciechanover A. (2000). Early work on the ubiquitin proteasome system, an interview with Aaron Ciechanover. Cell Death Differ Sep;12(9):1167-77. PMID 16094393
- Hershko A. (2005). Early work on the ubiquitin proteasome system, an interview with Avram Hershko. Cell Death Differ 12, 1158–1161. PMID 16094391
- Kopp F, Steiner R, Dahlmann B, Kuehn L, Reinauer H. (1986). Size and shape of the multicatalytic proteinase from rat skeletal muscle. Biochim Biophys Acta 872(3):253-60. PMID 3524688
- Löwe J, Stock D, Jap B, Zwickl P, Baumeister W, Huber R. (1995). Crystal structure of the 20S proteasome from the archaeon T. acidophilum at 3.4 Å resolution. Science 268, 533-539. PMID 7725097
- Groll M, Huber R, Potts BC (2006). Crystal structures of Salinosporamide A (NPI-0052) and B (NPI-0047) in complex with the 20S proteasome reveal important conséquences of beta-lactone ring opening and a mechanism for irreversible binding. J Am Chem Soc. 128(15):5136-41. PMID 16608349
- Wang J, Maldonado MA. (2006). The Ubiquitin-Proteasome System and Its Role in Inflammatory and Autoimmune Diseases. Cell Mol Immunol 3(4): 255. PMID 16978533
- Nandi D, Tahiliani P, Kumar A, Chandu D. (2006). The ubiquitin-proteasome system. J Biosci Mar;31(1):137-55. PMID 16595883
- Heinemeyer W, Fischer M, Krimmer T, Stachon U, Wolf DH. (1997). The Active Sites of the Eukaryotic 20S Proteasome and Their Involvement in Subunit Precursor Processing J Biol Chem 272(40):25200-25209. PMID 9312134
- Liu CW, Li X, Thompson D, Wooding K, Chang TL, Tang Z, Yu H, Thomas PJ, DeMartino GN. (2006). ATP binding and ATP hydrolysis play distinct roles in the function of 26S proteasome. Mol Cell Oct 6;24(1):39-50. PMID 17018291
- Lam YA, Lawson TG, Velayutham M, Zweier JL, Pickart CM. (2002). A proteasomal ATPase subunit recognizes the polyubiquitin degradation signal. Nature 416(6882):763-7. PMID 11961560
- Sharon M, Taverner T, Ambroggio XI, Deshaies RJ, Robinson CV. (2006). Structural Organization of the 19S Proteasome Lid: Insights from MS of Intact Complexes. PLoS Biol Aug;4(8):e267. PMID 16869714
- Higashitsuji H, Liu Y, Mayer RJ, Fujita J. (2005). The oncoprotein gankyrin negatively regulates both p53 and RB by enhancing proteasomal degradation. Cell Cycle 4(10):1335-7. PMID 16177571
- Forster A, Masters EI, Whitby FG, Robinson H, Hill CP. (2005). The 1.9 Å Structure of a Proteasome-11S Activator Complex and Implications for Proteasome-PAN/PA700 Interactions. Mol Cell May 27;18(5):589-99. PMID 15916965
- Witt S, Kwon YD, Sharon M, Felderer K, Beuttler M, Robinson CV, Baumeister W, Jap BK. (2006). Proteasome assembly triggers a switch required for active-site maturation. Structure Jul;14(7):1179–88. PMID 16843899
- Kruger E, Kloetzel PM, Enenkel C. (2001). 20S proteasome biogenesis. Biochimie Mar-Apr;83(3-4):289-93. PMID 11295488
- Gorbea C, Taillandier D, Rechsteiner M. (1999). Assembly of the regulatory complex of the 26S proteasome. Mol Biol Rep Apr;26(1-2):15-9. PMID 10363641
Voir aussi
Liens externes
- (en)[PDF]Ubiquitine-protéasome
- PLOS Primer: The Proteasome and the Delicate Balance between Destruction and Rescue
- (en)Protéasome de levure
- Portail de la biochimie
- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.