- Acide Ribo-Nucléique messager
-
Acide ribonucléique messager
 Pour les articles homonymes, voir messager.
Pour les articles homonymes, voir messager. Structure 3D de la molécule d'ARNm. Créé avec Rasmol.
Structure 3D de la molécule d'ARNm. Créé avec Rasmol.
L'acide ribonucléique messager, ARN messager ou ARNm est une copie de l'ADN utilisée comme intermédiaire par les cellules dans la synthèse des protéines. Le concept d'ARN messager a été émis puis démontré par Jacques Monod, François Jacob et leurs collaborateurs en 1960[1]. Toutefois, cette classe d'ARN a été isolée et étudiée en 1958 et appelé DNA-like ARN[2] par Volkin et Astrachan, mais sans que ceux-ci n'en comprennent le rôle biologique.
Sommaire
Principes
L'information génétique d'une cellule, qui gouverne tous les aspects de sa vie, est contenue dans son ADN. Cette information doit être traduite sous la forme de protéines, qui sont les molécules effectrices de l'organisme vivant. Plutôt que d'utiliser directement la matrice d'ADN pour synthétiser des protéines, l'évolution a établi une molécule intermédiaire, l'ARN messager, qui est une réplique d'une partie de l'ADN appelé gène. En effet, à un gène correspond en général une protéine.
Le passage par un intermédiaire ARNm permet aussi de réguler l'expression des gènes. Les besoins cellulaires pour une protéine donnée peuvent varier en fonction des conditions environnementales, du type cellulaire, du stade de développement, de l'âge de la cellule. Les ARNm sont des molécules labiles, dont la durée de vie est limitée, variant de quelques minutes à quelques heures. Leur production peut être adaptée par la cellule aux conditions spécifiques auxquelles la cellule est confrontée. Lorsqu'une protéine est nécessaire, la cellule transcrit l'ARNm correspondant. Lorsqu'à l'inverse elle n'en à plus besoin, la transcription du gène s'arrête et l'ARNm est progressivement dégradé par des ribonucléases (ou RNases). Ainsi la production de protéine peut être stimulée ou réprimée en fonction des besoins.
La régulation de la production d'une protéine à partir de son gène peut s'effectuer à plusieurs niveaux : par la régulation de la transcription de l'ADN en ARNm que l'on appelle le contrôle transcriptionnel, ou par le contrôle de la traduction de l'ARNm en protéine, que l'on appelle le contrôle traductionnel. La cellule peut donc « choisir » quelles parties de l'ADN seront transcrites et ainsi exprimées. Les différentes cellules expriment différentes parties du génome pour obtenir un phénotype différent selon la présence de facteurs de régulation.
Structure
Article détaillé : Structure de l'ARN.Comme tous les ARN, l'ARNm est un acide nucléique résultant de la polymérisation de ribonucléotides reliés par des liaisons phosphodiester. Comme le terme "ARN" l'indique, l'ose (ou plus familièrement "sucre") présent dans les ribonucléotides est le ribose. Les bases azotées présentes sur les ribonucléotides sont l'adénine (A) et l'uracile (U) complémentaires entre elles ainsi que la guanine (G) et la cytosine (C) complémentaires de même. L'ARNm emploie donc la base U, à la différence de l ADN qui utilise la base T[3]. Contrairement à l'ADN, l'ARNm est une molécule monocaténaire, c’est-à-dire formée d'un seul brin. Ceci n'empêche pas des repliements locaux complexes et très structurés de la molécule sur elle-même faisant intervenir la complémentarité entre bases azotées. On distingue deux niveaux d'organisation structurale de la molécule d'ARN : la structure secondaire, qui est définie par les appariements dit Watson-Crick entre bases, et la structure tertiaire, qui est une étape supplémentaire de repliement en trois dimensions de la molécule.
Dans le cas des ARNm, les structures secondaires peuvent jouer un rôle dans la régulation de l'expression des gènes, en modulant l'efficacité d'une ou plusieurs des étapes de transcription et de traduction. Par exemple, la présence de structures secondaires de la région 5' non-traduite peut influencer le recrutement du ribosome et donc l'efficacité de la traduction. Les structures secondaires peuvent également être des sites de liaisons pour des protéines qui modulent l'épissage ou la polyadénylation.
Phase de transcription
Article détaillé : Transcription (biologie).L'ARNm consiste en une copie sélective d'une région de l'ADN correspondant à un ou quelques gènes codant des protéines. L'opération de copie appelée transcription se déroule dans le noyau de la cellule chez les eucaryotes et dans le cytoplasme chez les procaryotes, le matériel génétique y résidant. La transcription a aussi lieu dans les organites semi-autonomes comme les mitochondries et les chloroplastes qui contiennent leur propre génome. Elle s'effectue en présence d'enzymes spécifiques appelées ARN polymérases et des nucléotides nécessaires à la synthèse de l'ARN.
L' ARN polymérase se fixe sur une séquence spécifique de l'ADN appelée promoteur de transcription, juste en amont du début de l'ARNm. Elle sépare alors les deux brins du duplexe d'ADN et crée ce qu'on appelle une bulle de transcription puis synthétise la molécule d'ARN en se servant du brin dit transcrit comme matrice. Un signal de terminaison, situé en aval du ou des gènes transcrit déclenche l'arrêt de la transcription, le détachement de l'ARN polymérase et la libération de l'ARNm terminé.
Chez les procaryotes, la molécule d'ARNm peut être directement traduite. Chez les eucaryotes (noyau et organites), la molécule synthétisée par les ARN polymérases est dans de nombreux cas de l'ARN pré-messager qui doit subir une maturation post-transcriptionnelle dans le noyau avant de pouvoir être traduit. En effet, les parties codantes (exons) chez les eucaryotes sont interrompues par des séquences non codantes (introns). Au cours de la maturation post-transcriptionnelle aura notamment lieu le phénomène d'épissage assurant la suture des séquences résultant de la transcription des exons alors que les séquences résultant de la transcription des introns seront retirées de l'ARN pré-messager.
Phase de maturation des transcrits ou modification post-transcriptionnelle
Article connexe : Modification post-transcriptionnelle.Après transcription de l'ADN en ARN, on obtient une structure moléculaire qui n'est pas encore "mature", et qui ne peut pas, dans la majorité des cas, être directement traduite en protéine. Ce manque de maturité vient du fait qu'on distingue dans sa structure des introns et des exons, dont seuls les exons sont codant pour la synthèse de la protéine et les introns doivent être épissés (retirés) avant que la phase de traduction puisse s'effectuer. Lors de cette maturation, il faudra aussi transformer légèrement cet acide ribonucléique pour le rendre plus résistant face à son environnement. Le but de cette maturation est de transformer cet ARN pré-messager (ARNpré-m) en ARN messager (ARNm). Plusieurs facteurs nucléaires interviennent dans ce processus :
- Les ribonucléoprotéines nucléaires hétérogènes : Après synthèse, le préARNm est entouré de protéines A, B et C nucléaires, la formation d'un complexe tétramérique tous les 600 nucléotides permet de maintenir les ARNpré-m sous forme monobrin et ainsi permettre la suite de la maturation.
- Les petits ARN nucléaires hétérogènes (ARNsn) : Ces ARNsn (small nuclear) sont transcrits de l'ADN par l'ARN polymérase III. Ils vont former un complexe avec un certain nombre de petites protéines basiques nucléaires. Le tout forme les ribonucléoprotéines (RNPsn, pour small nuclear), qui sont de petites particules ribonucléiques (notées de U1 à U6). Le rôle de ces protéines intervient au niveau de l'épissage des ARNpre-m ainsi que dans la méthylation de l'ARN pré ribosomique du nucléole (ARNpré-r).
Modifications en 5'
Article détaillé : Coiffe (biologie).Ces modifications se font au début de la synthèse d'ARNm (après 30 nucléotides). Toutes ces modifications se font en majorité par voie enzymatique.
- La première modification en 5' est l'ajout d'une coiffe à l'ARN cette coiffe est constituée de 7 méthyl-guanine (7mG). Cet ajout se fait par une enzyme, la guanylyl-transférase. Il y aura la formation d'un pont 5'-5' triphosphate dans le sens opposé de la synthèse normale. Tout ceci a pour but de faire barrière à l'action des 5' exonucléases, donc de protéger l'ARN de son environnement mais aussi un rôle dans l'épissage, dans le transport nucléocytoplasmique ainsi que dans la traduction.
- La seconde est une méthylation des riboses en 2'-OH.
- Lorsque le premier nucléotide transcrit est un A, on peut enfin avoir la formation de N6-méthyl-adénosine, par l'action d'une ARN méthylase qui est toutefois différente de l'ADN méthylase de maintenance.
Modifications en 3'
Article détaillé : Polyadénylation.Cette modification est une polyadénylation, autrement dit, il va y avoir ajout en 3' de l'ARN d'une queue de poly(A). Séquence de cette queue :
- séquence signal : 5'-AAUAAA-3'
- site d'hydrolyse : 5'-YA-3' où Y est une base pyrimidique(U ou C)
- région riche en -GU-
La séquence totale est donc de la forme: 5'-AAUAAA-..........-YA-..........-GU-5'
Il va ensuite y avoir l'intervention de l'ARN poly(A) polymérase qui va ajouter 250 résidus adényliques à cette queue. Cette queue a pour rôle la protection contre les ribonucléases et nucléolases hydrolysant en 3'-OH, ainsi qu'un rôle de stabilisation en vue de la traduction.
L'épissage
Article détaillé : épissage.L'épissage est un procédé de maturation des ARNpré-m se déroulant dans le nucléoplasme et favorisant l'excision des introns et la mise en continuité de différents exons. On peut donc remarquer que les gènes sont morcelés en: Exons : séquence traduite en protéine et en Introns qui ne seront pas traduits en protéine mais retiré lors de l'épissage.
Séquences remarquables des introns
- en 5', GU (1)
- en 3', poly(Y)11NC-AG (1et2)
- Entre 3' et 5', une séquence -CURAY- de branchement à 30 nucléotides de 3'
On utilise ici les codes à une lettre pour les combinaisons de bases pour représenter les variations possibles de bases.
Principe de l'épissage
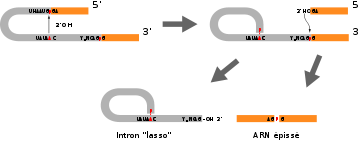
Il y a tout d'abord une hydrolyse en amont de G en 5' de l'intron, puis formation d'une liaison 2'5' phosphodiester de cette guanine avec l'Adénine de la séquence -CURAY-, ce qui conduit à une boucle intronique, communément appelé le lasso. On se retrouve alors avec un exon qui a son extrémité 3'OH libre. Il va ensuite y avoir une hydrolyse de la liaison phosphodiester en aval de G en 3', ce qui conduit à l'expulsion du lasso, les exons se retrouvent alors libres et vont être joints par une liaison phosphodiester.
Détail du mécanisme
Il y a intervention des snRNP dont la partie ARN s'apparie à la partie terminale des introns (commune pour tous les introns), la partie terminale de U1 se lie au site 5'-GU- et la partie terminale de U2 se lie au point de branchement A du -CURAY-. Puis, on assiste à une fixation de U4, U5, U6 sur U2 ce qui entraîne la formation du lasso
Phase de traduction
Article détaillé : Traduction.Les ARNm sont ensuite traduits en protéines par les ribosomes dans le cytoplasme. La petite sous-unité du ribosome (30S chez les bactéries ou 40S chez les eucaryotes) se fixe d'abord dans la région amont de l'ARNm et glisse jusqu'au codon de démarrage. Il recrute alors le premier ARN de transfert et la grande sous-unité du ribosome (50S chez les bactéries et 60S chez les eucaryotes) s'associe à l'ensemble. Le ribosome ainsi assemblé démarre a traduction de la protéine.
La traduction d'un ARNm comporte ainsi quatre grandes étapes successives :
- La fixation initiale de la petite sous-unité ribosomique sur l'ARN
- La formation du complexe de démarrage sur le premier codon de la phase ouverte de lecture
- L'élongation de la chaîne polypeptidique, à mesure que le ribosome avance sur l'ARNm
- La terminaison de la synthèse, lorsque le ribosome arrive sur le codon-stop. Cette phase permet la libération de la protéine terminée et la dissociation du ribosome de l'ARNm
Au cours de la phase d'élongation, l'ARNm traverse le ribosome au niveau d'un sillon de la petite sous-unité. Pendant ce procesus, un ou plusieurs ARN de transfert sont associés à l'ARNm au niveau de trois sites présents dans le ribosome.
- site A pour site Aminoacyl-ARNt. Ce site accueille les ARNt portant l'acide aminé qui va être ajouté à la chaîne. C'est au niveau du site A que s'effectue le décodage de l'information génétique, grâce à la reconnaissance de l'interaction entre le codon sur l'ARNm et l'anticodon sur l'ARNt
- site P : pour site Peptidyl-ARNt. Ce site accueille l'ARNt qui porte la chaîne peptidique déjà synthétisée. Ce dernier est toujours apparié à l'ARNm par son anticodon.
- site E ou site de sortie (en: exit). Ce site acceuille l'ARNt "nu" après transfert de la chaîne peptidique sur l'ARNt suivant, juste avant qu'il ne quitte le ribosome
Ces trois sites sont occupés séquentiellement par les ARNt au fur et à mesure de la progression du ribosome sur l'ARNm. A tout instant du processus de traduction, au plus deux de ces trois sites sont occupés simultanément : soit le site P et le site A, soit le site E et le site A.
Il existe une différence importante entre les ARNm eucaryotes et les ARNm bactériens : chez les eucaryotes, l'ARNm est systématiquement monocistronique, c'est à dire qu'il ne code qu'une seule chaîne protéique : le ribosome rentre via la coiffe 5' (unique aux eucaryotes). En revanche, l'ARNm procaryote est souvent polycistronique et contient donc plusieurs cistrons phases ouvertes de lecture. Il y a donc plusieurs sites de liaisons au ribosome.
ARN de transfert
Article détaillé : Acide ribonucléique de transfert.Les ARN de transfert ou ARNt sont des molécules adaptatrices qui font l'intermédiaire entre le codon porté par l'ARN messager et l'acide aminé qui sera incorporé dans la protéine en cours de synthèse par le ribosome. A l'un de leurs extrémités se trouve une boucle contenant un triplet de nucléotides, l'anticodon, qui est complémentaire du codon. Cet appariement codon-anticodon permet la reconnaissance de l'ARNt par le ribosome. Du coté opposé à l'anticodon, les ARNt portent un acide aminé attaché par une liaison ester à leur extrémité 3'-OH. Chaque cellule contient un ensemble d'ARNt différents, capables de s'apparier aux différents codons et spécifiques chacun d'un acide aminé donné. Leurs structures sont analogues, il adoptent une structure secondaire en forme de feuille de trèfle qui se replie en trois dimensions pour former une sorte de « L », avec l'anticodon d'un coté et l'acide aminé de l'autre.
Fidélité de la traduction
Article détaillé : Aminoacyl-ARNt synthétase.Le processus de traduction de l'ARNm en protéine a un taux d'erreur qui est de l'ordre de 10-4, soit un acide aminé incorrect sur 10000 acides aminés incorporés. La fidélité du mécanisme de traduction de l'ARNm par le ribosome repose sur deux étapes clés importantes. Il faut tout d'abord que chaque ARNt porte le bon acide aminé estérifié à son extrémité 3', une étape qui est réalisée par une famille d'enzyme, les aminoacyl-ARNt synthétases. Les aminoacyl-ARNt synthétases ont souvent une fonction de relecture ou proofreading, comme les ADN polymérases, qui leur permet de vérifier que le produit de leur réaction est bien correct et sinon de l'hydrolyser. L'autre étape critique est la vérification de l'interaction codon-anticodon par le ribosome, une étape qui est aussi soumise à un processus de relecture, assisté par le facteur d'élongation de la traduction EF-Tu.
Codage de l'information sur l'ARNm
- Initiation
Fixation d’ARN polymérase sur la séquence promoteur.
- Élongation
Synthèse d’ARN.
- Terminaison
La séquence de terminaison à la fin du gène arrête la transcription. L’ARN est libéré.
Voir aussi
Notes et références
Lien externe
- Portail de la biologie cellulaire et moléculaire
Catégorie : ARN
Wikimedia Foundation. 2010.