- Phosphorodiamidate morpholino oligo
-
Morpholino
En biologie moléculaire, un morpholino est un type de molécule utilisé pour modifier l'expression génétique. La technologie des oligonucléotides morpholinos est une technologie antisens utilisée pour bloquer l'accès d'autres molécules aux séquences spécifiques de certaines molécules d'acide nucléique. Ils peuvent bloquer l'accès des autres molécules à de petites régions (environ 25 bases) de la surface d'appariement des bases des acides ribonucléiques (ARN). Les morpholinos sont parfois référencés par l'appellation PMO, un acronyme anglophone signifiant "phosphorodiamidate morpholino oligo". Le mot "morpholino" peut apparaître dans d'autres noms chimiques. Il se rapporte à des produits chimiques contenant un cycle à six sous-unités morpholine; cet article discute seulement des oligonucléotides antisenses morpholinos.
En recherche biologique, les morpholinos sont habituellement utilisés en tant qu'outils pour la génétique inverse par inactivation de la fonction d'un gène. Ceci est accompli en empêchant les cellules de synthétiser une protéine [1] ciblée ou en modifiant l'épissage d'un pré-ARNm [2]. L’inactivation de l’expression des gènes est une méthode puissante pour étudier la fonction d’une protéine particulière. Dans le même ordre d’idée, induire l’épissage d’un exon spécifique peut aider à déterminer la fonction de la partie de la protéine codée par cet exon. Ces molécules ont été appliquées à l’étude de plusieurs organismes modèles, incluant la souris, le poisson-zèbre et le xénope [3].
Les morpholinos sont également développés en tant que thérapeutique pharmaceutique dirigée contre les organismes pathogènes tels que les bactéries [4] ou les virus [5] et pour l’amélioration des maladies génétiques [6]. Ces oligonucléotides synthétiques ont été conçus par le Dr James E. Summerton (Gene Tools, LLC) et développés en collaboration du Dr Dwight D. Weller (AVI BioPharma Inc.).
Sommaire
Structure
Les morpholinos sont des molécules synthétiques qui résultent d’un réarrangement de la structure naturelle des acides nucléiques [7]. Habituellement d’une longueur de 25 bases, ils se fixent aux séquences complémentaires des ARN par appariement de bases. Du point de vue de la structure, bien que les morpholinos possèdent des bases azotées classiques, ils diffèrent de l’ADN par le fait que ces bases azotées sont fixées sur des cycles morpholines au lieu de désoxyribose. Ces cycles morpholines sont liées entre-eux par des groupements phosphorodiamidates au lieu de groupements phosphates [7]. Il est plus facile de visualiser ceci en se référant à la figure 1 et en comparant les structures d’un brin d’ARN et d’un brin morpholino. Le remplacement des groupements phosphates anioniques par des groupements phosphorodiamidate non chargés élimine l’ionisation dans la gamme habituelle de pH physiologique, de sorte que les morpholinos dans les organismes ou les cellules sont des molécules non chargées. . Le squelette entier d’un morpholino est formé de ces sous-unités modifiées. Les morpholinos sont plus habituellement utilisés sous forme d’oligonucléotides monocaténaires, de sorte que les hétéroduplexes formés à partir d’un brin morpholino et un ADN complémentaire peut être utilisé en combination avec la livraison de réactifs cytosolique cationique [8].
Fonction
Contrairement à de nombreux types structurels antisens tels que les phosphorothioates ou les petits ARN interférants, les morpholinos ne dégradent pas les molécules d’ARN auxquelles ils se fixent. Les morpholinos agissent par encombrement stérique, en se fixant sur leur séquence cible sur un ARN ils se mettent sur le chemin de molécules qui en leur absence interagissent avec l’ARN[1].
Les oligonucléotides morpholinos sont souvent utilisés pour rechercher le rôle d’un ARNm spécifique transcrit chez l’embryon. Les biologiste du développement injectent les oligonucléotides morpholinos dans les œufs ou les embryons du poisson zèbre,[9], du xénope[10], le poussin[11] et l’oursin[12] produisant ainsi des embryons morphant. Avec un système de transfert cytoplasmique approprié, les morpholinos sont utilisés avec efficacité en culture cellulaire[8]
Les morpholinos sont développés en tant que produit pharmaceutique sous le nom de "NeuGene" par AVI BioPharma Inc. Ils ont été utilisés chez les mammifères allant de la souris[13] à l’être humain et certains sont couramment expérimentés dans les tests cliniques en tant que thérapie anticancer[14]
Expression normale ches les eucaryotes
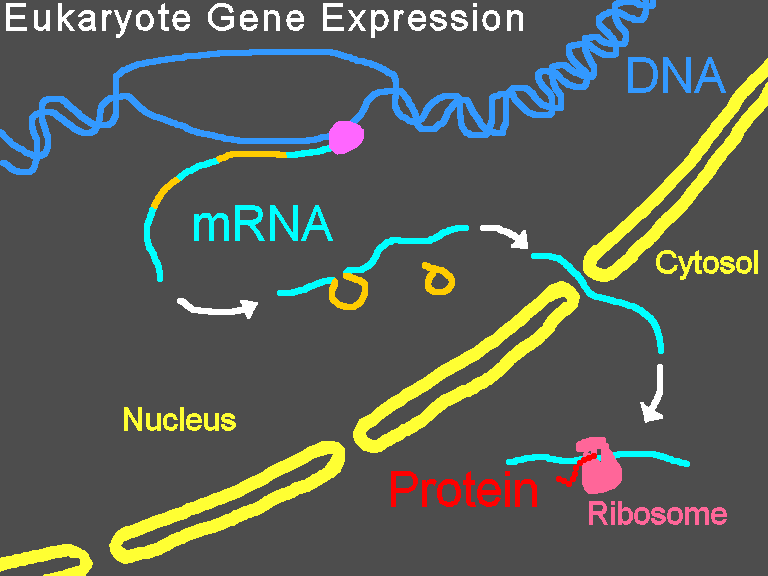 Expression des gènes eucaryotes sans intervention d’un morpholino
Expression des gènes eucaryotes sans intervention d’un morpholino
Chez les organismes eucaryotes, le pré-ARNm est transcrit dans le noyau, les introns sont épissés, puis l’ARNm mature est exporté du noyau vers le cytoplasme. Les petites sous-unités du ribosome commencent généralement par s’attacher à l’une des extrémités de l’ARNm et y sont rejointes par divers facteurs d’initiation, formant ainsi le complexe d’initiation. Le complexe d’initiation parcourt le brin d’ARNm jusqu’à ce qu’il rencontre le codon d’initiation, à ce moment la grande sous-unité du ribosome se fixe à la petite sous-unité. La traduction de la protéine débute. Ce processus entier est désigné sous l'appellation d'expression génétique. Il s’agit du processus par lequel l’information d’un gène, codé par la succession des bases azotées dans l’ADN, est converti en une structure protéique. Un morpholino peut modifier l’épissage ou bloquer la traduction selon la séquence en bases du morpholino.
Blocage de la traduction
 Traduction bloquée par un oligonucléotide morpholino
Traduction bloquée par un oligonucléotide morpholino
Liés à une région non traduite en 5' d’un ARN messager (ARNm), les morpholinos peuvent interférer avec la progression du complexe d’initiation ribosomique à partir de la coiffe en 5’ jusqu’au codon d’initiation. Cette inactivation de l’expression génétique empêche la traduction de la région codante du transcrit ciblée. Ceci est utile d’un point de vue expérimental lorsque qu’un chercheur souhaite connaître la fonction d’une protéine particulière. Les morpholinos fournissent un moyen commode pour inactiver l’expression de la protéine ciblée et étudier comment l’inactivation change la cellule ou l’organisme. Certains morpholinos inactivent l’expression si efficacement qu’après la dégradation des protéines préexistantes, les protéines ciblées deviennent indétectables par électrotransfert (p. ex. figure 1 dans :[15]).
Modification de l’épissage du préARNm
 Épissage bloqué par un oligomorpholino
Épissage bloqué par un oligomorpholino
Les morpholinos peuvent interférer avec les étapes de maturation du pré-ARNm, soit en empêchant l’accouplement du complexe des petites ribonucléoprotéines nucléaires (snRNP) avec leurs cibles au bord des introns sur le brin du pré-ARNm, soit en bloquant les adénines nucléophiles, prévenant de la sorte la formation d’épissage des structure en lasso ou en interférant avec l’attachement des protéines régulatrices d’épissage telles que les « splice silencers »[16] et les « splice enhancers »[17]. Empêcher l’attachement des snRNP U1 (sur le site donneur) ou U2/U5 (à moitié sur la polypyrimidine et à moitié sur le site accepteur) peut causer des modifications d’épissage, communément l’exclusion d’exons de la maturation de l’ARNm. Cibler certaines cibles d’épissage conduit à l’exclusion de l’intron, tandis que l’activation de sites d’épissage cachés peut conduire à des inclusions ou exclusions partielles[18]. Targets of U11/U12 snRNPs can also be blocked. Splice modification can be conveniently assayed by reverse-transcriptase polymerase chain reaction (RT-PCR) and is seen as a band shift after gel electrophoresis of RT-PCR products.[2]
Blocage des autres sites des ARNm
Les morpholinos ont été utilisés pour bloquer l’activité des micro ARN[19],[20] et pour bloquer l’activité des ribozymes [21]. Les fonctions des snRNP U2 et U12 ont été inhibée par les morpholinos[22]. Les morpholinos ciblant les séquences glissantes des ARNm dans les régions codantes peuvent induire un décalage des cadres de lecture.[23] Les activités des morpholinos contre cette variété de cibles suggère que les morpholinos peuvent être utilisés comme outils d’usage universel pour bloquer les interactions des protéines ou des acides nucléiques avec les ARNm.
Propriété intellectuelle
Gene Tools, LLC et AVI BioPharma Inc. sont des entreprises qui revendiquent la propriété intellectuelle sur différents aspects des oligonucléotides morpholinos.
Notes et références
- ↑ a et b J Summerton, « Morpholino Antisense Oligomers: The Case for an RNase-H Independent Structural Type. », dans Biochimica et Biophysica Acta, vol. 1489, 1999, p. 141-58 [{{Pubmed}} texte intégral]
- ↑ a et b BW Draper, « Inhibition of zebrafish fgf8 pre-mRNA splicing with morpholino oligos: A quantifiable method for gene knockdown. », dans Genesis, vol. 30, no 3, 2001, p. 154-6 [{{Pubmed}} texte intégral]
- ↑ Heasman J, « Morpholino oligos: making sense of antisense? », dans Dev. Biol., vol. 243, no 2, 2002, p. 209-14 [lien PMID]
- ↑ Geller BL, « Antibacterial antisense », dans Curr. Opin. Mol. Ther., vol. 7, no 2, 2005, p. 109-13 [lien PMID]
- ↑ TS Deas, « In vitro resistance selection and in vivo efficacy of morpholino oligomers against West Nile virus. », dans Antimicrob Agents Chemother., 2007, p. Epub ahead of print [{{Pubmed}} texte intégral]
- ↑ G McClorey, « Induced dystrophin exon skipping in human muscle explants. », dans Neuromuscul Disord., vol. 16, no 9-10, 2006, p. 583-90 [{{Pubmed}} texte intégral]
- ↑ a et b J Summerton, « Morpholino Antisense Oligomers: Design, Preparation and Properties. », dans Antisense & Nucleic Acid Drug Development, vol. 7*, 1997, p. 187-95 [{{Pubmed}} texte intégral]
- ↑ a et b PA Morcos, « Achieving efficient delivery of morpholino oligos in cultured cells. », dans Genesis, vol. 30, no 3, 2001, p. 94-102 [{{Pubmed}} texte intégral]
- ↑ A Nasevicius, « Effective targeted gene 'knockdown' in zebrafish. », dans Nature Genetics, vol. 26, no 2, 2000, p. 216 - 20 [{{Pubmed}} texte intégral]
- ↑ J Heasman, « Beta-catenin signaling activity dissected in the early Xenopus embryo: a novel antisense approach. », dans Developmental Biology, vol. 222, 2000, p. 124-34 [{{Pubmed}} texte intégral]
- ↑ R Kos, « The winged-helix transcription factor FoxD3 is important for establishing the neural crest lineage and repressing melanogenesis in avian embryos. », dans Development, vol. 128, no 8, 2001, p. 1467-79 [{{Pubmed}} texte intégral]
- ↑ EW Howard, « SpKrl: a direct target of (beta)-catenin regulation required for endoderm differentiation in sea urchin embryos. », dans Development, vol. 128, no 3, 2001, p. 365-75 [{{Pubmed}} texte intégral]
- ↑ SA Coonrod, « A morpholino phenocopy of the mouse MOS mutation. », dans Genesis, vol. 30, no 3, 2001, p. 198-200 [{{Pubmed}} texte intégral]
- ↑ Devi GR, Beer TM, Corless CL, Arora V, Weller DL, Iversen PL, « In vivo bioavailability and pharmacokinetics of a c-MYC antisense phosphorodiamidate morpholino oligomer, AVI-4126, in solid tumors », dans Clin. Cancer Res., vol. 11, no 10, 2005, p. 3930-8 [texte intégral lien PMID]
- ↑ I Stancheva, « A mutant form of MeCP2 protein associated with human Rett syndrome cannot be displaced from methylated DNA by notch in Xenopus embryos. », dans Mol Cell, vol. 12, no 2, 2003, p. 425-35 [{{Pubmed}} texte intégral]
- ↑ IG Bruno, « Correction of aberrant FGFR1 alternative RNA splicing through targeting of intronic regulatory elements. », dans Hum Mol Genet, vol. 3, no 20, 2004, p. 2409-20 [{{Pubmed}} texte intégral]
- ↑ F Vetrini, « Aberrant splicing in the ocular albinism type 1 gene (OA1/GPR143) is corrected in vitro by morpholino antisense oligonucleotides. », dans Hum Mutat, vol. 27, no 5, 2006, p. 420-6 [{{Pubmed}} texte intégral]
- ↑ PA Morcos, « Achieving targeted and quantifiable alteration of mRNA splicing with Morpholino oligos. », dans Biochem Biophys Res Commun, 2007 [{{Pubmed}} texte intégral]
- ↑ WP Kloosterman, « Substrate requirements for let-7 function in the developing zebrafish embryo. », dans Nucleic Acids Res, vol. 32, no 21, 2004, p. 6284-91 [{{Pubmed}} texte intégral]
- ↑ AS Flynt, « Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate. », dans Nature Genetics, vol. 39, 2007, p. 259-263
- ↑ L Yen, « Exogenous control of mammalian gene expression through modulation of RNA self-cleavage. », dans Nature, vol. 431, no 7007, 2004, p. 471-6 [{{Pubmed}} texte intégral]
- ↑ N Matter, « Targeted 'knockdown' of spliceosome function in mammalian cells. », dans Nucleic Acids Res, vol. 33, no 4, 2005, p. e41 [{{Pubmed}} texte intégral]
- ↑ MT Howard, « Efficient stimulation of site-specific ribosome frameshifting by antisense oligonucleotides. », dans RNA, vol. 10, no 10, 2004, p. 1653-61 [{{Pubmed}} texte intégral]
- Portail de la biologie
Catégories : Biologie moléculaire | Génétique | Morpholine | Acide nucléique
Wikimedia Foundation. 2010.

