- Empreinte génétique
-
 Cet article concerne le concept utilisé en identité judiciaire. Pour le phénomène de régulation génétique, voir Gène soumis à empreinte.
Cet article concerne le concept utilisé en identité judiciaire. Pour le phénomène de régulation génétique, voir Gène soumis à empreinte.Une empreinte génétique, ou profil génétique, est le résultat d'une analyse génétique, rendant possible l'identification d'une personne à partir d'une petite quantité de ses tissus biologiques (bulbe de cheveux, sang, salive, sperme).
L'empreinte génétique repose sur le fait suivant : bien que deux humains ont une large majorité de leur patrimoine génétique identique, un certain ensemble de séquences dans leur ADN reste spécifique à chaque individu (en raison du polymorphisme). Ce sont ces séquences spécifiques d'un individu que l'analyse d'empreinte génétique permet de comparer. Si un échantillon de cellules présente la même empreinte génétique qu'un individu, on peut soutenir que ces cellules proviennent de cet individu, ou de son éventuel jumeau monozygote.
Dans son acception initiale, l'expression empreinte génétique, de l'anglais « genetic fingerprint », est formée par analogie avec les empreintes digitales utilisées dans le cadre de l'identification des criminels, qui sont réputées propres à chaque individu.
Les empreintes génétiques sont utilisées en médecine légale pour identifier ou innocenter des suspects grâce à leur sang, leur salive, leurs poils ou leur sperme. Elles permettent également d'identifier des restes humains, de faire des test de paternité, d'organiser le don d'organe, d'étudier des populations d'animaux sauvages ou même de générer des hypothèses sur la diaspora humaine lors de la préhistoire (Bryan Sykes, Les Sept Filles d'Ève)
En raison du caractère sensible de cette information, les tests sont soumis à des contraintes légales. En France, le Comité consultatif national d'éthique a indiqué : « En matière civile et familiale, l'indisponibilité de l'identité civile et de la filiation, dont l'établissement ne requiert pas de preuve biologique en dehors d'un procès, la sécurité du lien parental dans l'intérêt primordial de l'enfant, l'équilibre et la paix des familles, justifient que la preuve biologique ne puisse être rapportée que sous le contrôle du juge, dans le cadre d'une action en justice relative à la filiation et juridiquement recevable »[1].
Sommaire
Historique
En 1985, Lord Alec Jeffreys (9 janvier 1950 - ), docteur britannique en génétique, découvre la méthode d'identification par l'ADN[2].
La technique est commercialisée en 1987.
En 1983, une jeune britannique âgée de 15 ans fut retrouvée morte. On crut tout d'abord à un meurtre par asphyxie, puis on décela sur son corps des marques de tortures et de viol. En 1986, tout près du lieu du premier meurtre, a été retrouvé un autre corps. Le mode opératoire était le même. En 1988, la méthode a été démocratisée, ce qui permit de disculper le suspect (qui deviendra alors le premier homme soumis à une disculpation dans l'histoire de la justice et de la criminalistique) et de retrouver le vrai coupable, qui fut condamné à la prison à vie[précision nécessaire].
Principes généraux
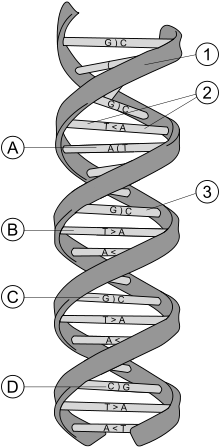
L'ADN est constitué de séquences de nucléotides parmi les quatre suivants : A (adénine), C (cytosine), T (thymine), G (guanine).
Il faut rappeler que les gènes permettent la fabrication des protéines. Mais il existe sur l'ADN des portions qui ne codent aucune protéine. Ce sont certaines d'entre elles, appelées les microsatellites et minisatellites, qui sont très variables selon les individus et permettent donc d'établir les empreintes génétiques.
Les microsatellites et minisatellites sont des séquences de nucléotides composées de répétitions de séquences plus petites. Il y a :
- les séquences répétées en tandem courtes, appelées aussi microsatellites ou STR, pour Short Tandem Repeats, en anglais. La plupart des séquences répétées sont des répétions de quatre nucléotides, mais les longueurs de deux à cinq bases sont aussi étudiés. Exemple :
CTGG CTGG CTGG CTGG CTGG CTGG - les minisatellites, ou VNTR, pour « Variable Number Tandem Repeats ». Les séquences répétées sont des répétions de 10 à 100 nucléotides. On regroupe parfois ces méthodes sous le nom « Multiple Loci VNTR Analysis » (Mlva).
Ces régions de l’ADN sont très polymorphes : en effet, le nombre de répétitions est variable pour chaque individu. Parce que les gens n’ont pas le même nombre de répétitions, ces régions de l’ADN permettent d'identifier les individus.
Les régions des chromosomes où se situent ces séquences (leurs locus) doivent être repérées, puis amplifiées par PCR : il s'agit de fabriquer de nombreuses copies de cet ADN pour que celui-ci soit « visible » à la fin de l'analyse. Les fragments d'ADN obtenus sont séparés et identifiés par électrophorèse. Ceci permet de connaître leur longueur et donc d'en déduire le nombre de répétitions.
Les deux méthodes de séparations les plus communes sont l’électrophorèse capillaire et électrophorèse sur gel.
Fiabilité
La grande force des systèmes basés sur les microsatellites est la fiabilité statistique de l’identification.
Le polymorphisme de chaque microsatellite est par lui-même très variable. Une version d'un même locus (ou allèle) peut avoir une fréquence comprise entre 5 et 20 % des individus. Un seul locus ne permet donc pas de désigner un individu précis. Il faut utiliser plusieurs locus.
En France[3] et aux États-Unis, on utilise couramment treize locus (régions de séquence répétée) pour une identification. Et comme chaque locus est composé d’une certaine séquence de répétition (microsatellite) et que le nombre de répétitions est parfaitement indépendant[réf. nécessaire] des répétitions sur les autres locus, les règles de statistiques peuvent être appliqués.
Par exemple, pour trois locus A, B et C, indépendants, et pour lesquels il existe plusieurs versions ( A1, A2, A3, et B1, B2, etc.) , on peut dire que Probabilité (A1, B2, C1)=Probabilité (A1) x Probabilité (B2,) x Probabilité (c1). Ainsi, pour 13 locus, la probabilité d’avoir deux séquences identiques pour deux individus différents non apparentés est estimée à 1 chance sur 1018 , ce qui est quasiment négligeable (très proche de zéro). Par conséquent, plus le nombre de microsatellites analysés est important, plus l’identification est fiable. Toutefois, dans le cas général, si on ne sait pas si les 2 individus sont apparentés, la probabilité monte à 1 chance sur 3x1012 (parce que 0,2 % de la population mondiale est constituée de jumeaux monozygotes).
Selon les pays, différents systèmes d’identification ADN basés sur la répétition des microsatellites sont utilisés. En Amérique du Nord (USA, Canada), la norme CODIS est la plus utilisée, alors qu’en Angleterre, c’est le SGM+. En France, les empreintes génétiques sont rassemblées dans le Fichier national automatisé des empreintes génétiques (FNAEG), destiné à l'origine à recueillir les empreintes des personnes condamnées pour infractions à caractère sexuel, mais dont l'usage s'est rapidement étendu à toutes sortes de délit, contenant en 2008 plus de 700 000 profils (soit près de 1 % de la population française) [4].
Mais, de toute manière, plusieurs zones de microsatellites sont communes aux différentes normes utilisées, ce qui permet la compatibilité entre elles.
Différentes techniques d'analyse
Il faut d'abord pratiquer l'extraction de l'ADN de l'échantillon de cellules. Il faut ensuite cibler et analyser les séquences répétées. La méthode par PCR est la plus utilisée.
PCR
Article détaillé : Réaction en chaîne par polymérase.Cette méthode présente beaucoup d'avantages :
- elle nécessite peu d'ADN, 1 à 2 nanogrammes;
- elle est rapide : une journée.
Avec l’invention de la réaction en chaîne par polymérase (appelée PCR en anglais), les tests ADN ont fait un bond en avant dans la précision et les possibilités d’analyse à partir d’un tout petit échantillon de départ.
La réaction en chaîne par polymérase permet de multiplier des séquences spécifiques d’ADN en jouant sur les propriétés d’hybridation et de dénaturation des brins complémentaires d’ADN en fonction de la température. Ces éléments permettent de contrôler l’activité enzymatique grâce à des transitions de température (assurées par un thermocycleur) répétées de manière cyclique (cf. réaction en chaîne).
L’un des principaux reproches fait à la méthode RFLP était la grande quantité et la qualité de l’échantillon d’ADN nécessaire.
Le développement de méthodes d’amplification permet l’analyse d’échantillons plus petits ou dégradés.Des méthodes comme le HLA-DQ alpha reverse sont devenues très populaire par leur facilité et la rapidité d’obtention des résultats, bien qu’elles soient moins précises que le RFLP. Par exemple, elles étaient inappropriées pour identifier différents ADN mélangés dans l’échantillon de départ comme pour ceux des fluides vaginaux des victimes de viols.
RFLP
Article détaillé : Polymorphisme de longueur des fragments de restriction.La Polymorphisme de longueur des fragments de restriction (RFLP) était une méthode longue et laborieuse (1 semaine) qui nécessite une grande quantité d'ADN de bonne qualité pour être utilisée. Elle a été supplantée par la méthode PCR.
L’analyse RFLP (« restriction fragment length polymorphism » : analyse du polymorphisme de longueur des fragments de restriction) est une des toutes premières techniques d’analyse ADN. Elle a été depuis complètement remplacée par des techniques plus récentes comme le Séquençage de l'ADN.
L’analyse RFLP utilise une enzyme de restriction pour découper l'ADN en fragments qui sont ensuite séparés en bandes par électrophorèse en gel d'agarose.
Ensuite, les bandes d'ADN sont transférées du gel d’électrophorèse sur une membrane nylon par une technique appelée Southern blot.
La membrane nylon recouverte des bandes de fragment d’ADN subit une irradiation qui fixe des séquences ADN spécifiques sur la membrane. l’ADN non fixé par les radiations est éliminé en lavant la membrane.
La membrane nylon avec les séquences d’ADN irradiés est alors placée sur un film sensible aux rayons X. Ce film est alors développé pour obtenir une image des bandes. L’image obtenue est appelé carte génétique ou « carte de restriction » d'une molécule d'ADN.
La carte de restriction donne l'ordre des sites de restriction le long de cette molécule, et la taille des fragments produits
En faisant plusieurs analyses sur différents morphismes, on arrive à un niveau de discrimination exploitable.
L'inconvénient principal de la méthode RFLP est que les tailles exactes des bandes sont inconnues et la comparaison à une échelle de poids moléculaire est purement qualitative.
Beaucoup de laboratoires ont développé des modèles décrivant ce qu'ils considéraient comme carte de restriction de référence, mais ils n'ont pas été standardisés et la prise des empreintes par RFLP a subi des attaques judiciaires (USA :Partie Civile vs. Castro 545 N.Y.S. 2e. 985 (Sup. Ct. 1989)).
AFLP
Article détaillé : Amplified fragment-length polymorphism.Une autre technique, AFLP ou amplified fragment-length polymorphism a été utilisée au début des années 1990. Cette technique est plus rapide que l’analyse RFLP et utilise la technique de la réaction en chaîne par polymérase pour répliquer les échantillons d’ADN.
On distingue les différentes allèles par le polymorphisme des Répétition en Tandem Polymorphe (variable number tandem repeat -VNTR- en anglais) séparées par électrophorèse sur gel polyacrylamide utilisant une échelle de mesure d’allèles (par opposition aux échelles de poids moléculaires).
Les bandes de migrations peuvent être visualisées en colorant le gel à l’argent. L’un des fragments ADN les plus utilisés pour l’analyse AFLP est le locus D1S80. Comme toutes méthodes basées sur la réaction en chaîne par polymérase, les ADN sévèrement dégradés ou en très petites quantités, peuvent produire des allèles marginaux répliqués en quantité insuffisante qui peuvent causer des erreurs. Par exemple, on pourrait conclure à tort à un ADN homozygote alors qu’il serait hétérozygote. De plus, comme l’analyse est réalisée sur gel, les Répétitions en Tandem Polymorphe de grands numéros peuvent s’accumuler en haut du gel rendant l’interprétation difficile.
L’AFLP est une méthode facilement automatisable qui permet la création de Classification phylogénétique sur la comparaison d’ADN individuel
À cause de son coût relativement modeste et de sa facilité de mise en place et d’exploitation, l’analyse AFLP reste répandue dans les laboratoires ayant peu de moyens ou peu d’exigences.
Analyse par amplification aléatoire d'ADN polymorphe
Article détaillé : Amplification aléatoire d'ADN polymorphe.Analyse de l'ADN mitochondrial
L'analyse de l'ADN mitochondrial (ADNmt) ne permet pas d'identifier à 100 % un individu, mais permet d'exclure une hypothèse, car cet ADN ne présente pas assez de variabilité dans les populations. En clair, comme plusieurs personnes peuvent présenter un même ADNmt, on ne pourra pas déterminer à laquelle de ces personnes appartient un échantillon d'ADNmt. On a environ 1 chance sur 2000 que deux personnes non apparentées présentent le même ADNmt.
Les médecins légistes étudient les sites HV1 et HV2 de l’ADNmt. Ce sont des séquences hypervariables (HV) dont les différentes versions sont nombreuses et avec des fréquences relativement faibles (quelques % de la population générale). Par conséquent ces marqueurs ne peuvent pas déterminer si un échantillon provient d'une unique personne. On pourra toujours proposer une probabilité que l'échantillon provienne d'un individu précis, mais cela ne constitue pas une preuve certaine. En revanche, si on obtient 2 ADNmt différents on peut être sûr qu'il ne s'agit pas de la même personne.
Avantage
Pour des échantillons fortement dégradés ou sur un cheveu sans bulbe, il est parfois impossible d'obtenir un profil complet de tous les microsatellites. Dans ce cas, on peut utiliser ADN mitochondrial car il y en a de nombreuses copies dans une cellule, tandis qu'il y a rarement plus de 1 ou 2 copies de l'ADN nucléaire.
Technique
Les médecins légistes amplifient les sites HV1 et HV2 de l’ADN mitochondrial (ADNmt) ; ils séquencent ensuite chaque région et comparent chaque nucléotide. On considère généralement que l’ADNmt testé n’est pas similaire à celui de référence lorsqu'une différence de plus de deux nucléotides est constatée.
Comme l’ADNmt est transmis uniquement par la mère, cette référence est l’ADNmt d'une des personnes de la lignée maternelle.
L’analyste doit faire preuve d’un peu de discernement car l’hétéroroplasmie et des différences poly-C peut brouiller des comparaisons de séquences.
ADNmt est utile dans la détermination d'identités peu claires, comme ceux de disparus si un parent maternellement lié peut être retrouvé.
L’analyse de l’ADNmt a été utilisé pour prouver l’imposture d’ Anna Anderson qui se prétendait être la princesse russe Anastasia Romanova.
Analyse du Chromosome Y
Il existe sur le Chromosome Y des régions hypervariables, nommées STRY.
Inconvénient
Le chromosome Y n'est présent que chez les individus de sexe masculin. Il ne pourra être effectué sur une femme.
Avantages
Le chromosome Y est transmis par le père, et l’analyse de ce chromosome permet de faire les tests de paternité, lorsque le père n'est plus vivant. Par exemple, on a pu éclaircir la controverse sur Sally Hemings en déterminant que Thomas Jefferson aurait bien eu un fils avec cette esclave.
Utilisation dans le domaine judiciaire
Les empreintes génétiques constituent un indice dont les implications en matière d'enquêtes criminelles et d'expertises légistes sont nombreuses. Elles peuvent ainsi confirmer la culpabilité d'un individu suspecté d'avoir commis un crime, si son empreinte génétique correspond à celle obtenue à partir de traces laissées sur les lieux du crime . À l'inverse, elles permettent également d'innocenter un suspect.
Généralement, les tribunaux reconnaissent la fiabilité des empreintes génétiques et acceptent les résultats de ces tests comme preuves lors des procès.
Article connexe : Base de données ADN.En France, ils alimentent le FNAEG, qui recensait en 2008 plus de 700 000 personnes, soit 1 % de la population. Aux Etats-Unis, la base Codis du FBI comportait en 2009 le profil de 6,6 millions de personnes[5]. Certaines polices (telle celle du Comté de Montgomery (Maryland)) veulent l'étendre à toutes les affaires de vol ou cambriolage, tandis que l'acceptation, par un référendum de 2004, de la Proposition 69 en Californie a étendu le recueil d'échantillons ADN (qui sont conservés par l'Etat tel quel) aux personnes non seulement condamnées, mais aussi simplement arrêtées sur la base d'une suspicion d'avoir commis un crime (felony), qui peut aller du vol au meurtre en passant par le piratage informatique ou l'émission de faux chèques[6]. La Californie a depuis été imitée par plus d'une dizaine d'Etats [5].
Aspects législatifs et juridiques
Discussion autour des preuves par test ADN
Au début des tests ADN comme preuves légales, les avocats argumentaient que compte tenu que la probabilité d’avoir la même carte génétique, était de 1 sur 5 millions, sur une population de 60 millions, il y avait statistiquement 12 personnes ayant les mêmes résultats aux tests ADN.
Ils considéraient donc que la probabilité n’était pas de 1 pour 5 millions mais 1 sur 12.
Cet argument ne peut être retenu à moins que le suspect ait été tiré au hasard dans la population. Or, généralement, le suspect a été appréhendé pour d’autres raisons que l’empreinte génétique.Quand les analyses RFLP furent mises au point, le risque théorique tomba à 1 sur 100 milliards. Néanmoins, le taux d’erreur de manipulation est sûrement supérieur au risque théorique car souvent les procédures actuelles ne reflètent pas la manière dont est calculée la probabilité de coïncidence.
Par exemple, la probabilité de coïncidence est fondée sur la probabilité que 2 marqueurs dans 2 échantillons provoquent une bande « exactement » au même endroit. Mais un laborantin pourrait conclure qu’un réseau de bandes similaires, mais pas totalement identiques, est en fait le même échantillon parce que ces différences sont dues à des imperfections de manipulation, comme une différence dans le gel d’agarose. Dans ce cas, le laborantin augmente le risque d’erreur en dérogeant aux critères de reconnaissance.
Des études récentes ont évalué le risque à un niveau suffisamment élevé pour être source de contestations[7].À cause de cela, des plafonds de probabilité arbitraires, supérieurs à ceux calculés, ont été mis sur les analyses RFLP. Aujourd’hui, les analyses RFLP sont largement inutilisées à cause de ces difficultés d’interprétations.
Les analyses STR ne souffrent pas de la subjectivité des laborantins, et permettent une bien meilleure précision de distinction d’ADN (de l’ordre de 1 sur 1013 avec une empreinte complète). Comme deux jumeaux monozygotes ont le même ADN, et que la probabilité d'avoir un tel jumeau pour un individu donné est de 1 sur 500, la précision de distinction d'individu n'est que de 1 sur 103.De toute manière, les analyse ADN doivent être prises en compte dans le cadre d’un contexte surtout si d’autres éléments le rendent moins fiables.
Ainsi, les contaminations d’échantillon par d’autres preuves (transfert secondaire) sont une source d’erreur souvent évoquée par les avocats de la défense.
Plus rarement, les cas de chimère génétique sont l’une des possibilités où la non concordance génétique disculpe à tort un suspect.
Si le test ADN est positif, on doit se poser les questions suivantes :- Peut il y avoir une coïncidence aléatoire ?
- Sinon, l’échantillon a-t-il été pollué ?
- Sinon, le suspect a-t-il laissé cet ADN au moment du crime ?
- Si oui, est-ce que cela signifie que l'accusé est coupable du crime ?
Erreurs dues aux analyses ADN
On peut voir la pertinence des analyses ADN comme preuves légales à la lumière d’affaires récentes, où les criminels ont laissé des échantillons de « faux » ADN sur les scènes de crimes.
Dans une affaire, un coupable a même dissimulé le faux ADN dans son propre corps : en 1992, le docteur John Schneeberger au Canada a violé une de ses patientes en la droguant auparavant. La Police a fait une prise de sang à Schneeberger et l’a comparé à l’ADN trouvé sur la scène du crime. À 3 reprises sans jamais constater une concordance entre les 2 ADN (celui du sperme et celui du sang). Il s’avère que le docteur s’était chirurgicalement implanté dans le bras un drain Penrose rempli d’un mélange d’anticoagulants et du sang de quelqu’un d’autre.
La question n'est donc pas celle d'une erreur technique (qui reste certes possible) mais bien du contournement des nouveaux moyens d'investigation par les criminels. C'est là que se situe la principale limite de la preuve génétique dans l'avenir : « la génétique ne devrait représenter dans l'esprit de tous qu'un élément, tout à fait nécessaire, mais non suffisant, pour le raisonnement. (...) le test ADN ne doit pas se substituer à l'enquête. Mais il faut aller plus loin. L'enquêteur doit impérativement, dans le cadre de son analyse et de ses réflexions, faire la critique de cette preuve génétique et imaginer les hypothèses où elle peut fausser l'interprétation des faits »[8].
Caractère aléatoire des analyses ADN
L’analyse des mêmes échantillons par différents laboratoires montrent que les résultats peuvent varier au point d’aboutir à des conclusions contradictoires[9].
Cas remarquables
Dans les années 1920, Anna Anderson déclarait qu’elle était la grande-duchesse Anatasia Romanova de Russie. Dans les années 1980, ses cendres de crémation ont été testées et montrent qu’elle n’avait aucun lien de parenté avec les membres restants de la lignée des Romanov.
La technique des empreintes génétiques mise au point par Alec Jeffreys a été utilisée pour la première fois en 1986 dans une affaire jugée par un Tribunal de Pennsylvanie[1].
En 1987, le boulanger anglais Colin Pitchfork fut le premier criminel confondu par une analyse ADN, à Leicester, la ville où furent mises au point les techniques d’analyse ADN.
Le 6 novembre 1987, en Floride, le violeur Tommie Lee Andrews a été la première personne aux États-Unis à être condamnée à 22 ans de prison sur la base d'une analyse ADN, pour le viol d’une femme au cours d’un cambriolage[10],[11].
En 1989, à Chicago, Gary Dotson a été le premier suspect innocenté par un test ADN.En 1992, un test ADN prouva que le tristement célèbre docteur nazi, Josef Mengele a été enterré au Brésil sous le nom de Wolfgang Gerhard
En 1994, O. J. Simpson fut acquitté alors que l’accusation était persuadée avoir présenté un dossier solide et attendait une condamnation. Notamment, les expertises ADN furent durement critiquées par la défense qui dénonça les procédures désordonnées ayant contaminé les échantillons.
En 1994, des tests ADN sur des poils de chat permirent de condamner un homme pour le meurtre de sa femme. Ce fut une première dans l’histoire de la médecine légale, de l’utilisation d’un ADN non humain pour identifier un criminel.
En 1998, le docteur Richards J Schmidt a été déclaré coupable de tentative de meurtre quand il fut montré un lien entre l’ADN de la souche virale VIH qu’il a été accusé d’avoir inoculé à sa compagne et celle d’un de ses patients atteint du Sida. Ce fut la première fois qu’un ADN viral a été utilisé comme pièce à conviction.
En 2003 le gallois Jeffrey Gafoor a été convaincu du meurtre de Lynette White quinze ans plus tôt. En réexaminant les éléments collectés et archivés de la scène du crime en 2000, des tests ADN utilisant les nouvelles techniques Mlva, permirent de confondre le neveu de la victime[12]. C’est le premier exemple connu d’identification d’un criminel par la « proximité ADN familial ».En mars 2003, Josiah Sutton a été relâché après avoir purgé 4 années de prison sur 12 pour sa condamnation pour agression sexuelle. Les échantillons d’ADN douteux furent retestés, ainsi que beaucoup d’autres, après la découverte de graves négligences au laboratoire de la Police de Houston
En juin 2005, avec une analyse ADN, Dennis Halstead, John Kogut et John Restivo ont gagné leur révision de procès suite à leur condamnation pour meurtre. Les trois hommes avaient déjà effectué dix-huit ans sur les trente ans de peine de prison.
Le procès Robert Pickton est remarquable car dans ce cas les analyses ADN ont été utilisées principalement pour identifier « les victimes » et dans de nombreux cas pour prouver leur existence.
Disculpation d'accusés et de condamnés
Earl Washington est libéré le 12 février 2001 après neuf années passées dans le couloir de la mort : il avait avoué en 1982 le viol et le meurtre d'une jeune femme et avait été condamné à la peine capitale bien qu'aucune preuve matérielle n'ait pu être retenue contre lui[13].
En décembre 2005, Robert Clark a été innocenté pour une agression sur une femme en 1981 à Atlanta après 24 ans de détention, parce qu’il avait mangé une pomme verte. M. Clark est la 164e personne aux États-Unis, la 5e dans l’État de Géorgie à être libéré après condamnation par un test ADN.
En juin 2009 la Cour suprême a refusé de faire du test ADN après condamnation un droit constitutionnel (District Attorney's Office v. Osborne).
Test ADN et loi française sur l'immigration
En 2007, en France, où une loi de bioéthique encadre les tests ADN, une polémique est soulevée lorsqu'un député propose d'utiliser les tests ADN pour prouver la filiation des étrangers souhaitant immigrer légalement dans le cadre du regroupement familial et de prouver par un test biologique, plutôt que par des documents parfois inexistants ou douteux.
Des parlementaires et intellectuels se sont opposés à cet amendement proposé par le député Thierry Mariani[14].
Une pétition lancée par l'hebdomadaire satirique Charlie-Hebdo et l'association SOS Racisme a rassemblé des dizaines de milliers de signatures[15], tandis que le chanteur Romeo Praly soutenait la pétition en mettant en musique un texte d'SOS-Racisme, du nom de « Touche pas à mon ADN ». Au sein du gouvernement, le ministre des Affaires étrangères Bernard Kouchner et Fadela Amara, la Secrétaire d'État chargée de la politique de la Ville, se sont opposés à cette loi ; Fadela Amara a déclaré, « y en a marre qu'on instrumentalise à chaque fois l'immigration, pour des raisons très précises. Je trouve ça dégueulasse ! ». Le philosophe et essayiste Pascal Bruckner l'a soutenue dans un plaidoyer condamnant cet amendement[16].
Les tests ADN sont utilisés dans onze pays européens, dont l’Allemagne, l’Italie, et le Royaume-Uni[17].
La proposition de loi fut remaniée pour n'autoriser les tests de filiation qu'à partir de l'ADN de la mère et ainsi éviter les révélations inadéquates d'adultères, et uniquement dans certains pays où l'état civil est défaillant.
Les dispositions qui prévoient les tests ADN mentionnés à l'article L.111-6 du CESEDA lorsque le décret d'application des conditions, mentionné au même article, sera paru, sont autorisées par l'article 226-28 du code pénal.
Notes et références
- OPECST - Empreintes génétiques, rapport n°3121
- A. J. Jeffreys, V. Wilson et S. W. Thein, « Hypervariable 'minisatellite' regions in human DNA », Nature, vol. 314, pp. 67–73 (1984).
- Rapport d'information sur le fichier national automatisé des empreintes génétiques, Assemblée Nationale, 2002
- Le fichier des empreintes génétiques contient 717.000 profils, Reuters, 23 avril 2008
- Jeffrey Rosen (en) (professeur à la faculté de droit de l'Université George Washington), Genetic Surveillance for All?, Slate, 17 mars 2009
- Laura K. Donohue, Proposition 69 could threaten privacy of DNA, SF Gate, 26 août 2004
- False result fear over DNA tests | Special reports | Guardian Unlimited.
- Jean-Hugues Matelly,«Influence de la preuve génétique dans l'enquête judiciaire», in 10 ans d'empreintes génétiques, La Documentation française, 2001.
- When a match is far from a lock ; L’expertise judiciaire ADN n’est pas infaillible.
- Gene Technology Page 14
- frontline: the case for innocence: the dna revolution: state and federal dna database laws examined
- Lynette White Case: How Forensics Caught the Cellophane Man
- (en)Washingtonpost.com
- « Test ADN : le oui, mais... du gouvernement », Le Figaro, 19 septembre 2007
- « Touche pas à mon ADN », site officiel de la pétition.
- « Fadela Amara a eu mille fois raison ! », Le Meilleur des Mondes, 10 octobre 2007
- Chloé Durand-Parenti et Cyriel Martin, « Test ADN et regroupement familial : l'amendement décrypté », Le Point, 14 septembre 2007
Voir aussi
Liens externes
- Dossier sur le site Planètegène
- Jean-Marc Manach, « ADN : quand les « experts » se trompent », LeMonde.fr, 10 décembre 2010
- Le parcours d'une empreinte génétique
Wikimedia Foundation. 2010.