- C3H7NO2Se
-
Sélénocystéine
Sélénocystéine 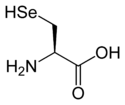
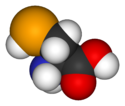
Structure de la L-sélénocystéineGénéral Nom IUPAC acide (R)-2-amino- 3-selanyl-propanoïque Synonymes U, Sec No CAS SMILES Propriétés chimiques Formule brute C3H7NO2Se [Isomères] Masse molaire 168,05 g∙mol-1
C 21,44 %, H 4,2 %, N 8,33 %, O 19,04 %, Se 46,99 %,Propriétés biochimiques Codons Codon-STOP UGA « opale » avec séquence d'insertion SecIS Unités du SI & CNTP, sauf indication contraire. La sélénocystéine est un acide aminé rare, qui entre dans la constitution de certaines protéines comme la glutathion peroxydase ou la formate déshydrogénase que l'on nomme sélénoprotéines. On dénombre actuellement trois gènes codant des sélénoprotéines chez E. coli.
D'après la nomenclature établie par l'IUPAC, la sélénocystéine s'abrège Sec pour le code à trois lettres et U pour le code à une lettre.
Contrairement à la plupart des acides aminés rares (n'appartenant pas à la série des 20 acides aminés communs aux polypeptides), la sélénocystéine n'est pas formée après la traduction de l'ARNm, mais incorporée directement lors de la synthèse de la chaîne polypeptidique par le ribosome.
Sommaire
Synthèse
La sélénocystéine dérive métaboliquement de la sérine, convertie en sélénocystéine alors que le résidu d'acide aminé est déjà fixé à son ARN de transfert. Structurellement, en revanche, elle est équivalente à une molécule de cystéine dont on a remplacé l'atome de soufre par du sélénium.
L'enzyme qui effectue la conversion de sérine en sélénocystéine sur l'ARNt est la sélénocystéine synthase [1]. La sélenocystéine synthase utilise le sélénophosphate (PO3Se3-) comme donneur activé de sélénium, produit par la sélénophosphate synthétase à partir de séléniure.
Incorporation de la sélénocystéine dans les protéines
L'incorporation de la sélénocystéine dans les protéines est un processus complexe, car il n'existe pas à proprement parler de codon spécifique pour cet acide aminé. Dans l'ARN messager, à la position correspondant à la sélénocystéine dans la protéine, on trouve un codon UGA, qui est normalement un codon-STOP (appelé « opale »), signal d'arrêt de la traduction. Il y a cependant un ARNt spécifique permettant l'incorporation de cet acide aminé dans la chaîne polypeptidique en formation, dont l'anticodon est complémentaire de UGA. La discrimination entre le codon UGA codant la sélenocystéine du codon-STOP UGA nécessite un mécanisme spécifique :
- Il existe une machinerie protéique spécifique qui s'associe à l'ARNt sélénocysteine pour diriger son incorporation au site A du ribosome pendant l'étape d'élongation
- l'ARN messager porte un élément de structure secondaire spécifique appelé élément SecIS (pour Selenocystein Insertion Sequence) qui est impliqué dans le recrutement du complexe protéine/ARNt chargé. Le sélénocystéinyl-ARNt est alors incorporé au site A du ribosome.
Chez les bactéries, le système est composé du facteur protéique SelB, homologue au facteur d'élongation "classique" EF-Tu capable de reconnaitre les 20 autres aminoacyl-ARNt. SelB est homologue à EF-Tu sur sa partie N-terminale et de ce fait est capable de lier le GTP, l'ARNt chargé et d'interagir avec le ribosome. Il possède de plus une extension C-terminale composée de quatre domaines en "winged-helix" se liant avec une très forte affinité (nM) à l'élément SecIS, une épingle-à-cheveux, ou « tige-boucle », dans l'ARNm située juste après le codon UGA. La SecIS est composée de 40 nucléotides de séquence variable mais dont certains éléments invariants et nécessaires à l'interaction avec SelB ont pu être mis en évidence.
Chez les eucaryotes et les archées, le mécanisme est légèrement différent. Chez les eucaryotes, deux protéines en complexe, mSelB et SBP2, jouent respectivement le rôle de facteur d'élongation et de liaison aux éléments SecIS. Ces derniers sont également différents en séquence et en taille des SecIS procaryotes, et se situent le plus souvent en 3' hors de la partie codante du gène.
Notes et références
Articles liés
- Portail de la biochimie
- Portail de la chimie
Catégories : Composé du sélénium | Acide aminé
Wikimedia Foundation. 2010.
