- Liaison isopeptidique
-
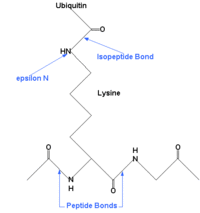
Une liaison isopeptidique est une liaison peptidique qui ne se trouve pas dans la chaine principale de la protéine. Ce lien se forme entre les fonctions « acide carboxylique » et « groupe aminé » d'une chaine latérale (figure 1.).
Des liaisons isopeptidiques peuvent se créer entre l'amine de la chaîne latérale de la lysine et les groupes carboxyles de la chaine latérale du glutamate ou de l'aspartate[1].
La formation de la liaison peut être catalysée par voie enzymatique, comme dans le cas de la liaison formée entre la lysine et de la glutamine, qui est catalysée par des transglutaminases, ou peut se former spontanément comme observé dans la formation du capside du bactériophage HK97[2] et dans les pili des bactéries Gram positives[3].
La formation spontanée d'une liaison isopeptidique nécessite la présence d'un autre résidu, l'acide glutamique, qui catalyse la formation de liaisons induites par proximité.
Un exemple d'un petit peptide contenant une liaison isopeptidique est le glutathion, qui a un lien entre la chaîne latérale d'un résidu de glutamate et le groupe amine d'un résidu cystéine.
Un exemple d'une protéine impliquée dans la liaison isopeptidique est l'ubiquitine, qui se lie à d'autres protéines par une liaison entre la glycine C-terminale de l'ubiquitine et une chaîne latérale de la lysine de la protéine substrat.
Les liaisons isopeptidiques font partie des modifications post-transcriptionnelles courantes.
Applications
Récemment, des chercheurs ont exploité la formation spontanée d'une liaison isopeptidique afin de créer une nouvelle balise peptidique appelée isopeptag.
L'isopeptag peut spontanément et irréversiblement réagir avec son partenaire de liaison à travers une liaison isopeptidique covalente.
Des applications de ce nouvel outil moléculaire peuvent être : le ciblage de protéines in vivo, la microscopie à fluorescence, et le développement de nouvelles architectures de protéines pour la biologie synthétique[4].
Notes et références
- Stryer, Lubert. Biochemistry 4th Edition, 942-943 (1995)
- Wikoff, W. R. et al. Topologically linked protein rings in the bacteriophage HK97 capsid. Science 289, 2129-2133 (2000).
- Kang, H. J., Coulibaly, F., Clow, F., Proft, T. & Baker, E. N. Stabilizing isopeptide bonds revealed in gram-positive bacterial pilus structure. Science 318, 1625-1628 (2007).
- Zakeri,B. and Howarth,M. (2010). Spontaneous intermolecular amide bond formation between side chains for irreversible peptide targeting. J. Am. Chem. Soc. 132, 4526-4527.[(en) Résumé]
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Isopeptide bond » (voir la liste des auteurs)
Wikimedia Foundation. 2010.