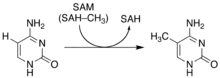- ADN méthyltransférase
-
Les ADN méthyltransférases sont des enzyme qui catalyse le transfert de groupes méthyle d'une S-adénosylméthionine sur des résidus cytosine(majoritairement en position C5) ou adénine (généralement en N6) (majoritairement en position C5). Les cytosines méthylées les plus connues précédent un résidu guanine dans ce que l'on appel les îlots CpG[1],[2]. Environ 60 à 90 % de ces régions sont méthylées dans le génome des mammifères[3].
La méthylation de l’ADN est un phénomène épigénétique, c’est-à-dire qu’il regroupe l’ensemble des modifications chimiques se surimposant à la séquence d’ADN sans aucune altération des séquences nucléotidiques. Elle est transmise au cours des divisions cellulaires et peut être réversible[4].
Sommaire
Méthylation chez les eucaryotes
ADN méthyltransférases impliquées dans la méthylation des îlots CpG (DNMTs)
Chez les mammifères, Il existe deux types de méthylation : méthylation de maintenance et méthylation de novo réalisées par trois classes générales d’enzymes[3].
- DNMT 1 permet la maintenance des profils de méthylation au cours des divisions cellulaires en reproduisant sur le brin néosynthétisé, lors de la réplication de l’ADN, la méthylation lue sur le brin matrice.
- DNMT 2 a une fonction inconnue, identifiée par homologie de séquence avec DNMT1.
- DNMT 3 est responsable de la méthylation de novo en ajoutant des groupements méthyles sur les 2 brins d’ADN[5].
- DNMT 3a est impliquée dans la méthylation des promoteurs de l'expression des gènes.
- DNMT 3b est impliquée dans la méthylation des séquences entourant les centromères[6].
Mécanisme
Les DNMTs présentent une partie N-terminale qui interagit avec l'ADN et d'autres protéines, et une partie C-terminale portant l'activité catalytique. Une séquence NLS (nuclear localization signal) présente sur l’enzyme permet de cibler le noyau de la cellule contenant l’ADN[7]. La S-adénosyl méthionine (SAM) est utilisée comme substrat donneur de groupements méthyles (-CH3), qui s’ajouteront à la place d'un atome d'hydrogène sur les cytosines en position C5[5],[8].
Rôles
La méthylation de l’ADN est une marque répressive, c’est-à-dire que la méthylation d’un îlot CpG est incompatible avec l’activité transcriptionnelle[2]. La méthylation de l’ADN peut inactiver la transcription par la présence d’un groupement méthyle qui empêche l’interaction entre le gèneet les facteurs de transcription[8]. Dans de nombreux gènes, les régions promotrices à l'extrémité 5' sont riches en dinucléotides CpG. La méthylation du promoteur peut attirer des protéines spécifiques qui reconnaissent l'ADN méthylé dont la plus connue est MecP2 (méthyl cytosine-binding protein (protéine de liaison à la méthylcytosine)). Lorsqu'elles interagissent avec le gène, elles vont recruter des complexes multiprotéiques contenant notamment des histones désacétylases. Ces désacétylases vont entrainer une désacétylation des nucléosomes ce qui abouti à une compaction de la fibre de chromatine. Cela entraine un décrochage du complexe de transcription, dont le site de fixation n’est plus accessible, aboutissant donc à une répression transcriptionnelle réduisant le gène au silence[9]. L’expression de nombreux gènes dépend donc fortement de la conformation de la chromatine, c’est-à-dire de son degré de condensation[10]. De plus, la méthylation de l’ADN intervient dans l’empreinte génomique parentale. Il s’agit d’une marque épigénétique correspondant à l’origine paternelle et maternelle[11],[12]. Ces modifications sont spécifiques de certaines séquences formant ainsi des « centres d’empreintes » ou des « éléments de contrôle de l’empreinte »[4],[13].
Chez les champignons
L’extinction génique, par l’inhibition de l’élongation de la transcription, est due à la seule méthylation des cytosines de la région codante[14].
Chez les plantes
L’initiation de la transcription serait inhibée par la méthylation des cytosines du promoteur[14]. Le pourcentage de méthylcytosine varie de 5% à 37% suggérant ainsi une grande variabilité d’une espèce à l’autre. La méthylation de l’ADN chez les plantes à un rôle important dans leur développement et leurs réponses à l’environnement [15].
Méthylation chez bactéries
La méthylation de l’ADN est un mécanisme cellulaire permettant la protection de l’intégrité du génome. Il s’agit d’un mécanisme très ancien, inhibant l’activation d’éléments mobiles (viral ou bactérien) ayant pénétré dans la cellule[9]. Les ADN étrangers introduits dans la cellule possèdent une méthylation différente par rapport à l’ADN de la cellule bactérienne réceptrice. Ces ADN étrangers sont donc dégradés par des enzymes de restriction sensibles à la méthylation et spécifique d’une séquence, tandis que l’ADN bactérien n’est pas reconnu par ces enzymes[2]. Par conséquent, la méthylation de l'ADN agit comme un système immunitaire primitif, permettant aux bactéries de se protéger contre l'infection par un bactériophage. Les bactéries possèdent la faculté de méthyler la position N6 des adénines situés dans les séquences CATG par l'action des N-adénine méthyltransférase ou "Méthylases Dam"; ou les cytosine en première position des séquences CCAGG et CCTGG par l'action des "Méthylases Dcm"[16]. Il existe une troisième classe de méthylase bactérienne, les méthylases EcoKI qui méthyle l'adénine en gras des séquences AACAGTGC et GCACAGTT.
Dérèglement de la méthylation de l’ADN dans le cancer et autres pathologies
Une altération de la méthylation de l’ADN peut favoriser l’apparition d’une pathologie. En effet, les cellules cancéreuses peuvent présenter des aspects inhabituels de la méthylation de l'ADN.
- L’hypométhylation peut avoir des effets observés dans les maladies cardio-vasculaires, les affections hépatiques, la dépression, le vieillissement prématuré et dans le développement du cancer à travers différents mécanismes.
- L’hyperméthylation affecte les gènes suppresseurs de tumeurs qui sont par conséquent anormalement réduit au silence. Ils ne peuvent donc plus freiner la croissance tumorale[7].
Notes et références
- (fr) Fabian Vandermeers, Richard Kettmann et Luc Willems, « Implication des modifications épigénétiques dans les cancers : développement de nouvelles approches thérapeutiques », dans Biotechnologie, Agronomie, Société et Environnement, 12 (2) 2008, p. 211-218.
- (fr) Michaël Weber, « Profils de méthylation de l’ADN dans les cellules normales et cancéreuses », dans Médecine/Sciences n° 8-9, vol. 24, août-septembre 2008 ; consulté le 21/04/2011
- News-medical.net ; DNA methylation in mammals
- (fr) J. M. Dupont, 4èmes Journées du Collège Universitaire et Hospitalier des Histologistes, Embryologistes, Cytologistes et Cytogénéticiens, Dourdan, Mars 1999.
- Fabian Vandermeers, Richard Kettmann et Luc Willems - Implication des modifications épigénétiques dans les cancers : développement de nouvelles approches thérapeutiques -volume 12 (2008) - Gembloux Agricultural University – Unité de Biologie cellulaire et moléculaire
- Thierry Forné - MODIFICATIONS EPIGENETIQUES - Institut de Génétique Moléculaire – CNRS Montpellier
- (fr) Jérôme Torrisani, Frédéric Lopez, « Méthylation de l'ADN et régulation épigénétique des cancers », institut Louis-Bugnard.
- Dr UME - METHYLATION DE L’ADN - S&P Laboratories – consulté le 21/04/2011
- (fr) Dr Jean-Michel DUPONT - Empreinte parentale - consulté le 19/04/2011
- (fr) François Fucks - Médecine/sciences 2003, vol.19 , 477-80 / p478– consulté le 21/04/2011
- (fr) Anne Gabory, Luisa Dandolo - Épigénétique et développement: l’empreinte parentale - MEDECINE/SCIENCES n° 4, vol. 21, avril 2005 – p.390-5 – consulté le 18/04/2011
- (fr) Pr. Manuel MARK - EMPREINTE GÉNOMIQUE PARENTALE - Institut d’Embryologie, Faculté de Médecine et Hôpital Universitaire de Strasbourg – consulté le 20/04/2011.
- (fr) Thomas-D Pollard, William-C Earnshaw, « Biologie cellulaire », Elsevier/Masson, juillet 2004, p. 230-231. (ISBN 2-84299-571-6)
- (fr) Hervé Lalucque, « Études de dégénérescence cellulaire causées par des éléments non-conventionels infectieux dans le champignon filamentaux Podospora Anseria », Université de Parise XI UFR scientifique d’Orsay, 18 mars 2002.
- (fr) F. Brignolas, A. Delaunay, C. Hébrard, C. Joseph, C. Lafon-Placette, S. Maury, « Rôle de la méthylation de l’ADN dans la plasticité développementale des plantes et recherche d’épiallèles en vue d’une sélection agronomique », Laboratoire de Biologie des Ligneux et des Grandes Cultures, consulté le 20/04/2011.
- (en) DNA methylation in bacteria - News-medical.net – consulté le 18/05/2011.
Catégorie :- EC 2.1.1
Wikimedia Foundation. 2010.