- Phospholipase C
-
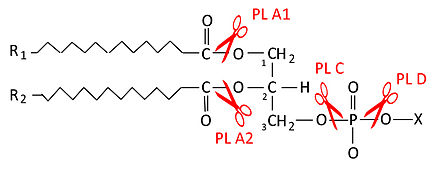
Les phospholipases sont des enzymes hydrolysant les liaisons esters des phospholipides. Il existe quatre liaisons ester dans un phospholipide. On distingue donc plusieurs enzymes selon leur site d’action sur la molécule.
La phospholipase C est l'une d'entre elles. Elle intervient sur la fonction ester liant le glycérol et le phosphate, libérant un diglycéride et un phosphoalcool. In vivo, les phospholipases C catalysent l’hydrolyse du phosphatidylinositol diphosphate membranaire (PIP2) en diglycéride et inositol-1-4-5-triphosphate (IP3).
Il existe plusieurs espèces de phospholipase C :
- La phospholipase Cβ, qui est activée par les récepteurs couplés aux protéines G ;
- La phospholipase Cγ, qui est activée par des récepteurs enzyme à activité tyrosine kinase ;
- La phospholipase Cδ, qui est activée par l’IP3 ;
- La phospholipase Cε, qui est activée par la petite protéine G Rap
Les phospholipases Cβ participent à la production de l’IP3, alors que les phospholipases Cδ sont activées par l’IP3.
Sommaire
La phospholipase Cβ
La phospholipase Cβ est une enzyme transmembranaire. Suite à son activation par un récepteur couplé aux protéines G, l’hydrolyse du PIP2 membranaire provoque une augmentation de la concentration intracellulaire du second messager IP3. La fixation d'IP3 sur ses récepteurs induit l’activation des canaux calciques des citernes du réticulum endoplasmique, provoquant la sortie du calcium de ces réserves et donc une augmentation de la concentration cytosolique des ions Ca2+.
Pour une faible concentration de Ca2+ libérée on observe un phénomène de coopérativité positive entraînant une augmentation de l’affinité des autres sites pour IP3. Pour de fortes concentrations en calcium libérées on a un phénomène de Feed Back négatif qui se traduit par une baisse de l’affinité de l’IP3 pour ses récepteurs (mode de régulation en cloche).
4 molécules d’IP3 doivent se fixer pour que le récepteur soit fonctionnel, il s'agit donc de récepteur tétramérique à 4 sites de fixation.
L’IP3 peut par ailleurs subir une nouvelle phosphorylation pour donner l’inositol-1-3-4-5-tétraphosphate (IP4) qui est capable d’agir spécifiquement sur des canaux calcique de la membrane plasmique permettant l’entrée du calcium extracellulaire dans la cellule.
Au final, on assiste à une augmentation rapide et importante de la concentration cytosolique de calcium : c’est le signal calcique. Ce signal calcique entraîne l’activation de diverses protéines calcium dépendantes, comme la calmoduline, elles-mêmes capables d’activer des protéines kinase qui induisent divers effets cellulaires par des cascades de phosphorylations. Le calcium permet également le transport ou la translocation d’une protéine kinase C du cytoplasme où elle est inactive, vers la membrane plasmique où elle est rendue active par les diglycérides issus de l’hydrolyse du PIP2. Ces diglycérides peuvent aussi être hydrolysés par une diglycéride lipase qui libère de l’acide arachidonique (20:4).
Une fois que les effets sont induits, l’IP3 peut être dégradé dans le cycle des phosphoinositides.
Les stocks de calcium intracellulaire sont reconstitués grâce à l’activation d’une pompe : SERCA (Sarcoplasmic endoplasmic REticulum CAlcium pump). L’activation de SERCA permet l’entrée de Ca2+ à l’intérieur des stocks intracellulaires. Elle fonctionne en présence d’ATP contre un gradient de concentration (système de transport actif).
La phospholipase Cγ
La phospholipase Cγ diffère de la phospholipase Cβ de par sa structure. Elle possède deux domaine SH2, 1 domaine SH3 et 1 domaine PH (= Pleckstrin Homology). La phospholipase Cβ ne possède pas de domaine SH2.
La phospholipase Cδ
La phospholipase Cε
Voir aussi
Articles connexes
Wikimedia Foundation. 2010.