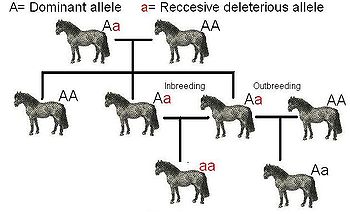
- Dépression endogamique
-
La dépression endogamique, ou dépression de consanguinité, désigne la réduction de la valeur sélective d'une population donnée liée à la reproduction entre individus apparentés. La reproduction entre individus étroitement apparentés, ou consanguinité, fait apparaître plus de caractères récessifs délétères. Plus les géniteurs sont apparentés et plus les descendants portent des gènes homozygotes délétères, donnant des individus inaptes. Un autre mécanisme responsable de la réduction de la valeur sélective est la superdominance d'allèles hétérozygotes dans une population à nombreux génotypes homozygotes, même s'ils ne sont pas délétères. En fait on ne sait pas lequel de ces deux mécanismes est le plus important. En général, les populations ayant une forte variation génétique ne souffrent pas de la dépression endogamique, qui est souvent le résultat d'un goulot d'étranglement génétique. Elle semble présente chez la plupart des groupes d'organismes, mais peut-être davantage chez les espèces hermaphrodites, plus particulièrement chez les plantes (qui sont en majorité hermaphrodites).
Sommaire
Dépression endogamique et sélection naturelle
La sélection naturelle ne peut pas éliminer tous les gènes délétères récessifs d'une population pour plusieurs raisons. D'abord, ces gènes apparaissent constamment par mutations au sein d'une population. Ensuite, dans une population où la consanguinité est fréquente, la plupart des descendants possèdent certains caractères délétères, si bien que peu d'entre eux seront plus aptes que les autres à la survie. Les différents caractères délétères n'affecteront cependant probablement pas de la même manière la reproduction. Un caractère récessif particulièrement désavantageux chez un individu homozygote récessif s'éliminera vraisemblablement de lui-même, limitant ainsi l'expression de son phénotype. Enfin, les allèles récessifs délétères seront « masqués » chez les individus hétérozygotes, et ces derniers ne subiront pas la pression sélective (effet de dominance).
Gestion de la dépression endogamique
L'introduction de nouveaux gènes d'une population différente peut contrer la dépression endogamique. Des populations différentes ont des caractères délétères différents, et par conséquent la plupart des locus ne seront pas homozygotes chez les descendants. Cela est connu sous le nom d'hétérosis, pratiqué dans les zoos pour prévenir l'homozygotie. Toutefois, mélanger deux populations différentes peut conduire à des caractères polygéniques indésirables de dépression exogamique.
Chez l'homme
Bien que les cas de forte consanguinité soient rares chez l'homme, il existe certaines formes apparentes de dépression endogamiques. Comme chez les animaux, ces phénomènes tendent à se produire dans des populations rurales, isolées, coupées à un certain point des autres zones de civilisation. Un exemple notoire est constitué par la tribu Vadoma du Zimbabwe occidental, dans laquelle le caractère d'ectrodactylie (présence de deux orteils seulement) paraît lié à un pool génique réduit.
Espèces non sujettes à la dépression endogamique
Exemples de taxons non sujets à une dépression endogamique significative malgré leur taille de population effective très faible :
Animaux
- Miro des Chatham
- Crécerelle de Maurice
- Rat-taupe nu (mammifère à structure reproductive eusociale et faible variation génétique[1],[2])
- Stegodyphus dumicola et certaines autres araignées sociales (vivant dans des colonies fortement consanguines)
- Thai Ridgeback (race de chien)
- Chien chanteur (sous-espèce de chien sauvage)
Plantes
- Le pissenlit se reproduit de manière asexuée par apomixie.
- Schiedea verticillata
- Toromiro
Voir aussi
Articles connexes
- Hétérosis
- Population minimum viable
- Génétique des populations
- Vadoma
- Évolution insulaire
Bibliographie
- Charlesworth, D. & Charlesworth, B. (1987). Inbreeding depression and its evolutionary consequences. Ann. Rev. Ecol. @st., 18, 237458
- Lande, R. & Barrowclough, G. F. (1987). Effective population size, genetic variation, and their use in population management. In Viable populations for conservation, ed. M. E. Soulr. Cambridge University Press, Cambridge, pp. 87-123
Notes, sources et références
- (en) Faulkes CG, Abbott DH, OBrien HP, Lau L, Roy MR, Wayne RK, Bruford MW, « Micro- and macrogeographical genetic structure of colonies of naked mole-rats Heterocephalus glaber », dans MOLECULAR ECOLOGY, BLACKWELL SCIENCE LTD, P O BOX 88, OSNEY MEAD, OXFORD, OXON, ENGLAND OX2 0NE, vol. 6, no 7, juillet 1997, p. 615-628 (ISSN 0962-1083) :
« "Individuals within colonies were genetically almost monomorphic, sharing the same mtDNA control region haplotype and having coefficients of band sharing estimated from DNA fingerprints ranging from 0.93 to 0.99." »
- (en) Stanton Braude, « Dispersal and new colony formation in wild naked mole-rats: evidence against inbreeding as the system of mating », dans Behavioral Ecology, Oxford University Press, vol. 11, no 1, 2000, p. 7-12 [texte intégral]
(en) Cet article est partiellement ou en totalité issu de l’article en anglais intitulé « Inbreeding depression » (voir la liste des auteurs)- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.