- Épissage
-
Chez les eucaryotes (organismes dont les cellules possèdent un noyau), l’épissage est un processus par lequel les ARN transcrits à partir de l'ADN génomique peuvent subir des étapes de coupure et ligature qui conduisent à la suppression de certaines régions dans l’ARN final. Les segments conservés s’appellent des exons et ceux qui sont éliminés s’appellent des introns.
Les gènes sont donc constitués d’une suite d’exons et d’introns alternés ; ceci s’observe principalement dans les gènes codant des protéines, mais aussi dans certains gènes d’ARN non codants, comme ceux d’ARNt.
Lors de la transcription de gènes codant des protéines, un pré-ARNm est synthétisé puis est épissé dans le noyau de la cellule pour donner lieu à l’ARNm dit mature. L’ARNm mature, constitué des seuls exons, est alors exporté vers le cytoplasme pour être traduit en protéine.
L’épissage est assuré par un ensemble de complexes ribonucléoprotéiques appelé collectivement splicéosome (épissage se disant splicing en anglais). Chaque complexe, appelé snRNP pour Small Nuclear Ribonucleoprotein, contient un ARN et plusieurs protéines.
Il existe également des introns appelés auto-épissables ou auto-catalytiques, c’est-à-dire capable de s’exciser sans intervention d’un splicéosome, dans les mitochondries, les plastes et certaines bactéries. Cependant, au moins dans les mitochondries et les chloroplastes, certains de ces introns nécessitent l’intervention de protéines nucléaires.
Le mécanisme catalytique du splicéosome est encore inconnu, mais par analogie avec le fonctionnement du ribosome, on pense que c'est l’ARN qui est catalytique (et donc que le splicéosome est un ribozyme).
Sommaire
Structure des introns
Les jonctions intron/exon contiennent des séquences nucléotidiques caractéristiques qui sont conservées. Ces séquences sont reconnues par le spliceosome. L'intron contient en plus une séquence interne, dite boite de branchement. Cette boite de branchement comporte une adénosine qui joue un rôle central dans le processus d'épissage.
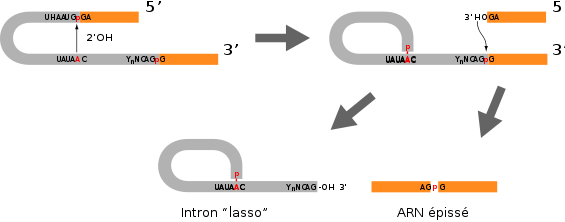
 Schéma de l'organisation des jonctions exon-intron-exon. Les exons amont et aval sont en orange et l'intron est en gris. Les nucléotides conservés sont indiqués.
Schéma de l'organisation des jonctions exon-intron-exon. Les exons amont et aval sont en orange et l'intron est en gris. Les nucléotides conservés sont indiqués.L'épissage proprement dit s'effectue en deux temps, on a tout d'abord une attaque nucléophile du 2'-OH du ribose de l'adénosine de la boite de branchement sur le phosphate de la jonction exon-intron en 5'. Après cette coupure, le 3'-OH libéré au niveau de l'exon amont attaque le phosphate de la jonction intron-exon en aval. Les produits de cette réaction sont d'une part les deux exons ligaturés correctement et d'autre part, l'intron cyclisé au niveau de l'adénosine de la boite de branchement. En raison de sa forme particulière, cette forme de l'intron est appelée lasso (en: lariat). Le lasso est finalement ouvert par une enzyme de débranchement afin de pouvoir être recyclé
Mécanisme d'épissage
Les snRNP
Les snRNP ou small nuclear ribonucleoproteins sont des composants du spliceosome présents dans le noyau des cellules eucaryotes. Le processus d'épissage canonique fait appel à cinq snRNP, appelés U1, U2, U4, U5 et U6. Chacun de ces snRNP est composé d'un ARN, appelé ARNpn, qui adopte une structure secondaire et tertiaire spécifique, et de plusieurs proteines. Parmi les protéines associées, certaines sont communes à tous les snRNP et d'autres sont spécifiques de chacun d'entre eux. Les proteines communes sont appelées protéines Sm, elles sont au nombre de sept et s'associent pour former un anneau heptamérique entourant un segment du snARN.
Les protéines spécifiques sont en nombre variable, il y en a par exemple trois pour U1: U1A, U1C et U1-70K.
Processus d'épissage
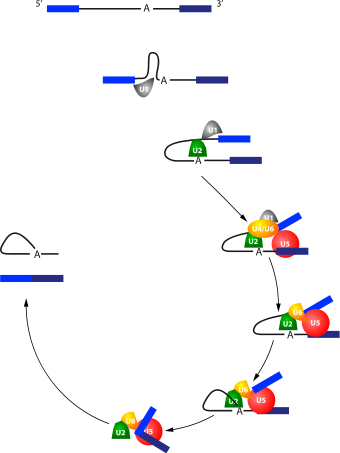 Mécanisme d'épissage d'un intron par le spliceosome. Les deux exons sont représentés par des barres bleues et l'intron par un trait noir.
Mécanisme d'épissage d'un intron par le spliceosome. Les deux exons sont représentés par des barres bleues et l'intron par un trait noir.
Le processus d'épissage est un mécanisme dynamique qui suit une succession précise d'évènements au cours de laquelle les différents snRNP s'assemblent et se désassemblent du spliceosome. La séquence est la suivante :
- U1 s'associe à la jonction 5' de l'intron.
- U2 s'associe à la boite de branchement.
- U4 associé à U6 rapproche U1 et U2, réalisant ainsi un pont entre la jonction 5' de l'intron et la boite de branchement.
- U5 s'associe à son tour et rapproche les bords 3' et 5' des exons à suturer.
- U4 et U1 quittent le complexe.
- Le 2'-OH du A de la boite de branchement coupe la jonction 5' de l'intron.
- Le 3'-OH du nucléotide en 3' de l'exon amont coupe l'autre jonction.
- L'ARNm épissé et l'intron en "lasso" sont libérés.
Association des composants du spliceosome
La plupart des interactions entre l'ARNm et le les snRNP passent par des appariements de base entre portions de séquences nucléotidiques complémentaires. Ainsi par exemple le snARN U1 contient une séquence complémentaire de la séquence consensus retrouvée à la jonction exon-intron en 5', ce qui lui permet de se lier à cette région des pré-ARN messagers. Il existe également des appariements ARN-ARN entre les différents snARN.
Introns autocatalytiques
Chez certains protozoaires (organismes eucaryotes uni-cellulaires), certains ARN introniques n'ont pas besoin de spliceosomes, ils suffisent à catalyser les réactions d'épissage ("self-splicing") sans apport de protéines extérieures. Ce phénomène relativement rare concerne les introns formant un ribozyme. On trouve deux sortes d'introns autocatalytiques : Les groupes 1 et 2.
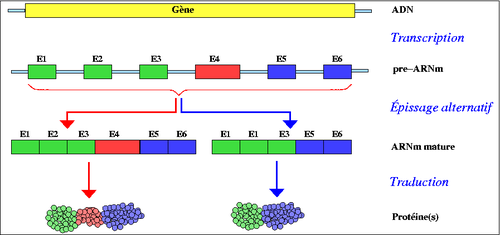
Épissage alternatif
Chez les eucaryotes uniquement, c'est une étape nécessaire pour rendre un ARN messager viable à la traduction. Cette étape consiste en la délétion (excision) des introns ou des exons qui ne seront pas nécessaires au codage de la protéine, en l'addition d'une coiffe méthylée en 5' et d'une queue poly(A) en 3'. Ces derniers éléments seront nécessaires au déplacement de l'ARN messager du noyau vers le cytoplasme, où il sera traduit par les ribosomes, ainsi qu'à sa stabilité, puisqu'il ne sera plus attaquable par les ARNs nucléosidases.
En effet le spliceosome reconnaît des signaux d'épissage, comme pour un signal radio, ces signaux d'épissage sont plus ou moins forts, ce qui implique que le spliceosome les reconnait plus ou moins bien.
Ces signaux sont simplement des séquences spécifiques de nucléotides.
Les signaux faibles sont appelés « signaux d'épissage alternatif », ils vont permettre à un pre-ARNm d'être épissé en plusieurs ARNm matures. Par opposition les signaux forts sont appelés « signaux constitutifs ».
C'est ainsi qu'un gène peut coder plusieurs protéines. L'épissage alternatif joue un rôle très important dans le développement des cellules, l'organisation des tissus et même dans le développement d'un individu. (par exemple : le gène Slx pour la différentiation du sexe chez la drosophile). « On pense qu'au moins 70 % des quelque 30 000 gènes qui composent le génome humain subissent un épissage alternatif et que, en moyenne, un gène donne naissance à 4 variants issus d'un tel épissage, pouvant donner naissance à environ 100 000 protéines différentes de par leur séquence et, du coup, leurs activités. » [1],[2] Et le dogme « 1 gène pour 1 protéine » n'est plus.
Dans certains cas extrêmes, l'épissage alternatif permet à un seul gène de coder plus de protéines que tous les autres réunis. C'est le cas pour Dscam chez la drosophile qui peut coder jusqu'à 38016 ARN messagers différents[3].
L'épissage alternatif permet donc à un gène de coder plusieurs ARNm différents. Il peut se faire de plusieurs façons.
- Le lasso ne comprend pas toujours un intron seul. Pour un gène contenant un grand nombre d'exons, le lasso formé peut comprendre 2 introns et 1 exon au lieu d'un seul intron. On aura alors des exons non-représentés dans l'ARNm mature, et donc dans la protéine.
- On peut trouver plusieurs promoteurs au sein d'un même gène. De ce fait, les ARNm matures ne contiennent que les exons en aval du promoteur, ce qui entraine la traduction de plusieurs protéines différentes selon le promoteur utilisé lors de l'épissage.
- On peut trouver des séquences polyA (séquence de résidus adénine qui protège l'ARNm des exonucléases) à l'intérieur de l'ARNm en plus de la séquence polyA que l'on trouve à l'extrémité 3' de l'ARNm. Selon la séquence polyA qui sera coupé avant traduction, on aura donc des protéines traduites différentes.
L'épissage alternatif est un processus important de régulation de l'expression des gènes. Deux conséquences de l'épissage alternatif y concourent :
La première est connue sous les initiales NMD Nonsense mediated decay : lors de l'épissage alternatif, l'ajout ou le retrait d'exon, ou bien encore la modification de la longueur de tel ou tel exon (par rapport à l'ARNm "canonique") peut entraîner un décalage du cadre de lecture (les nucléotides sont lus trois par trois par le ribosome lors de la traduction), et de ce fait causer l'apparition prématurée d'un codon STOP, ce qu'on appelle une mutation non-sens. Les ARNm ainsi formés sont reconnus par un ensemble de protéines et dégradés avant que la traduction puisse s'opérer. Ce processus n'est pas seulement un mécanisme de "contrôle qualité" mais permet aussi, en augmentant le taux des épissages alternatifs producteurs de codons STOP prématurés, de diminuer le nombre de protéines traduites. La seconde conséquence est tout simplement le fait qu'un changement dans la séquence de l'ARNm se répercute sur celle de la protéine et donc potentiellement sur ses capacités physico-chimiques, et permet de réguler sa fonction. Par exemple : la protéine CD45 joue un rôle dans l'activation des lymphocytes T lors d'une réponse immunitaire à une infection. Lorsque les lymphocytes T sont au repos et donc potentiellement activables, la forme la plus longue de la protéine est exprimée (elle comporte la totalité des neuf exons). Lorsque les lymphocytes T viennent d'être activés, les ARNm de CD45 sont épissés alternativement et les exons 4,5,6 sont excisés. La forme de CD45 ainsi produite est donc plus courte et ne peut pas jouer son rôle d'activateur. Ceci empêche les lymphocytes T de recevoir un signal d'activation trop longtemps et donc de déclencher une réponse immunitaire disproportionnée.
Notes et références
- Un article sur le net. Epissage alternatif. (2009). ExonHit Therapeutics.
- Hewetson A, Wright-Pastusek AE, Helmer RA, Wesley KA, Chilton BS: Conservation of inter-protein sites in RUSH and RFBP, an ATP11B isoform. Mol Cell Endocrinol. (2008) Sep 24;292(1-2):79-86. PMID: 18584949
- AM Celotto, BR Graveley, Genetics, Vol. 159, No. 2. (October 2001), pp. 599-608
- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.