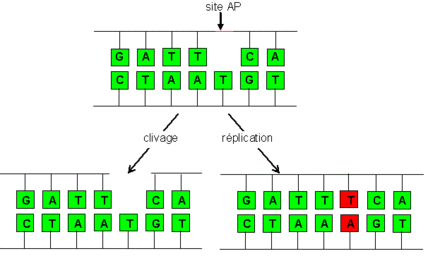
- Site AP
-
Un site AP (site apurinique/apyrimidique), ou site abasique, est un emplacement de l'ADN où la base (purine ou pyrimidine) est manquante. En termes de séquence génétique, c'est donc un site "vacant".
Sommaire
Origines
Les sites AP apparaissent spontanément par dépurination ou plus rarement par dépyrimidination (hydrolyse), au rythme d'environ 10000 par jour et par cellule. Dans chaque cellule, on en trouve en permanence entre 50 000 et 200 000, ce taux variant d'un tissu à l'autre[1],[2].
Ils constituent également le premier stade de la réparation par excision de base (REB) de l'ADN. Dans ce processus, une enzyme spécialisées (une glycosylase) identifie une base abimée et clive le lien N-glycosidique pour libérer la base: le site abasique ainsi créé peut (et doit) ensuite être réparé par d'autres enzymes pour reconstituer la séquence génétique initiale[3].
Réparation
Normalement, les sites AP isolés sont réparés via l'action successives des enzymes du système REB (principalement endonucléase AP, ADN polymérase β, puis ADN ligase)[4].
Les sites AP peuvent également apparaître suite à une irradiation. Dans ce cas on observe souvent des "grappes", c'est-à-dire plusieurs sites AP localisés côte-à-côte. Ces grappes sont très difficiles à réparer, et peuvent persister pendant plusieurs jours[5].
Effets délétères
En l'absence de réparation, les sites AP évoluent en quelques jours vers une rupture simple brin de l'ADN (effet clastogène)[6].
Les sites AP ont par ailleurs un très fort effet mutagène : au moment de la réplication, les ADN polymérase n'ont aucun moyen de savoir quelle base placer en face de ces sites vacants, et vont "choisir" une base arbitrairement. Le plus souvent, les polymérases vont insérer préférentiellement une adénine (la "règle du A"), de sorte que le site abasique sera remplacé par une thymine[7],[8].
Enfin, les semble qu'un site AP puisse dans certaines conditions engendrer une réticulation inter-brin (en anglais, (en) interstrand cross-link), ce qui pourrait mener à la mort de la cellule (effet cytotoxique)[9].
Voir aussi
Articles connexes
Bibliographie
- Réplication et réparation de l'ADN, Cours du Master Recherche "Biologie structurale et bio-informatique", Université Louis Pasteur Strasbourg, Département Intégrité du Génome, UMR CNRS-7175,
- Maintenance et variations du matériel génétique, Pr. J. Lunardi, Cours de Biochimie et Biologie Moléculaire, Université Joseph Fourier de Grenoble
- Altération et réparation de l’ADN, Cours de Licence Sciences et Technologie, Module de Biologie moléculaire 2, Université Paris 12 Val-de-Marne
Notes et références
- Nakamura 1999, Endogenous apurinic/apyrimidinic sites in genomic DNA of mammalian tissues, Cancer Research 59:2522-2526
- De Bont 1999, Endogenous DNA damage in humans: a review of quantitative data, Mutagenesis 19:169-185
- Wyatt 1999, 3-Methyladenine DNA glycosylases: structure, function, and biological importance, BioEssays 21:668–676
- Strauss, Abasic Site Repair in Higher Eukaryotes, DNA Damage and Repair, Vol III: Advances from phage to humans, Chapitre 3, Humana Press, 2001
- Georgakilas 2004, Processing of bistranded abasic DNA clusters in gamma-irradiated human hematopoietic cells, Nucleic Acids Research 32:5609–5620
- Jan Vijg, Aging of the Genome: The dual role of DNA in life and death, Oxford University Press, 2007
- Eoff 2010, Mechanistic Studies with DNA Polymerases Reveal Complex Outcomes following Bypass of DNA Damage, Journal of Nucleic Acids, Article ID 830473
- Obeid 2010, Replication through an abasic DNA lesion: structural basis for adenine selectivity, The EMBO Journal 29, 1738 - 1747
- Dutta 2007, Interstrand cross-links generated by abasic sites in duplex DNA, Journal of the American Chemical Society 129:1852-1852
Liens externes
- Reactome - a curated knowledgebase of biological pathways
- REPAIRtoire, A database of DNA repair pathways
- KEGG PATHWAY Database, section génétique
- Portail de la biologie cellulaire et moléculaire
- Portail de la microbiologie
Wikimedia Foundation. 2010.