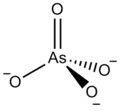
- GFAJ-1
-
 GFAJ-1
GFAJ-1
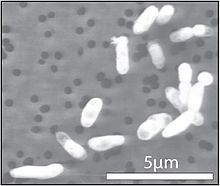
Cellules de bactérie GFAJ-1 sur un milieu
contenant de l'arsenic, mais pas de phosphoreClassification Règne Bacteria Embranchement Proteobacteria Classe Gamma Proteobacteria Ordre Oceanospirillales Famille Halomonadaceae Genre Halomonas GFAJ-1[1] est un bacille extrêmophile de la famille Halomonadaceae et du genre Halomonas[2], isolé dans le lac Mono en Californie. Cette souche, lorsqu'elle est cultivée dans un milieu dépourvu de phosphore, serait[3] capable d'utiliser l'arsenic, habituellement toxique, pour le remplacer dans la formation des nucléotides.
Sommaire
Description
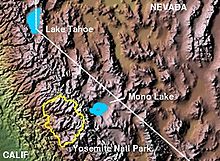 Carte montrant la localisation du Lac Mono, à l'est du parc national de Yosemite
Carte montrant la localisation du Lac Mono, à l'est du parc national de Yosemite
Cette souche bactérienne, GFAJ-1, a été découverte par la géomicrobiologiste Felisa Wolfe-Simon, une biologiste de la NASA travaillant à l’United States Geological Survey à Menlo Park, en Californie. Elle a été prélevée en 2009 dans des sédiments que Wolfe-Simon et ses collègues avaient recueilli sur les rives du lac Mono. Ce lac est très riche en sels et fortement alcalin et possède aussi l'une des plus fortes concentrations en arsenic au monde (200 µmol⋅L-1). Ce bacille a démontré pouvoir croître dans un milieu dépourvu de phosphore, en métabolisant l'arsenic présent dans le milieu sous forme d'ion arséniate[4].
Phylogénie
Phylogénie de la souche GFAJ-1 Thioalkalimicrobium cyclicum
Thioalkalivibrio jannaschii
Escherichia coli strain O157:H7
Halomonas alkaliphila
Halomonas venusta strain NBSL13
GFAJ-1
Halomonas sp. GTW
Halomonas sp. G27
Halomonas sp. DH77
Halomonas sp. mp3
Halomonas sp. IB-O18
Halomonas sp. ML-185
Phylogénie de la souche GFAJ-1 et de quelques bactéries proches sur la base de la séquence du gène codant l'ARN ribosomique 16S[5] Selon les résultats du séquençage du gène codant l'ARN ribosomique 16S, la bactérie GFAJ-1 fait partie du genre Halomonas. Ces bactéries sont des bactéries halophiles, également connues pour tolérer des niveaux élevés d'arsenic, mais GFAJ-1 peut faire mieux. Lorsqu'elle est privée de phosphore, elle peut utiliser l'arsenic à la place, et continuer de croître.
Détails sur l'article du 2 décembre 2010 dans le journal Science[6]
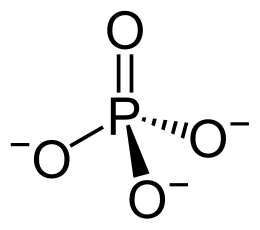 Ion phosphate
Ion phosphate
Le jeudi 2 décembre 2010 à 14 h EST, la découverte de cette souche est annoncée lors d'une conférence de presse de la NASA, au même moment le journal Science publie en ligne l'article scientifique décrivant l'isolement de cette souche et les tests effectués.
Isolement d'une souche pure
La souche GFAJ-1 a été isolée sur les rives du lac Mono, un lac hypersalin, hyperalcalin (pH 10) et possédant une concentration moyenne en arsenic de 200 μmol⋅l-1. La capacité à utiliser l'arsenic à la place du phosphore a été mesurée en incubant des sédiments du lac Mono dans un milieu de culture minimum défini. Celui-ci ne contenait pas de phosphore ni de supplément nutritif comme l'extrait de levure (un supplément habituellement utilisé pour les cultures de micro-organismes). Par la suite, le milieu de culture a été enrichi en arsenic sous la forme d'ion arséniate, d'une concentration initiale de 100 μmol⋅l-1 à une concentration finale de 500 µmol⋅l-1. Sept dilutions décimales en série[note 1] ont été effectuées sur une période de trois mois, résultant en une dilution finale de l'inoculum originel d'un facteur 107 [5]. Les dilutions successives permettent de diminuer au maximum la présence de phosphore, la concentration résiduelle en phosphore a été mesurée à 3,1 μmol⋅l-1 (contamination du milieu initial, des sels constituant le milieu et de la décomposition des organismes déjà présent dans le milieu de départ).
Le sixième transfert, sur les sept dilutions, manifestait un taux de croissance de 0,1 DO par jour (unité de densité optique mesurée au spectrophotomètre à une longueur d'onde de 680 nm). Cette culture fut utilisée pour inoculer un milieu solide (même milieu plus de l'agar). Deux types morphologiques de colonies furent observés, prélevés sur ce milieu solide ils furent mis en culture liquide sans phosphore. La culture manifestant la croissance la plus rapide fut retenue pour le reste des expériences : à ce stade les chercheurs avaient obtenu une culture pure d'une souche encore non identifiée. Cette souche pouvait être maintenue en culture dans un milieu dépourvu de phosphore, mais contenant 40 mmol⋅l-1 d'arséniate et 10 mmol⋅l-1 de glucose.
Identification de la souche pure
La souche bactérienne GFAJ-1 a été identifiée par séquençage du gène codant pour l'ARN ribosomique 16S. L'analyse phylogénétique de cette séquence indique que cette bactérie appartient à la famille des Halomonadaceae, et probablement au genre Halomonas.
Métabolisme de la souche
Lorsqu'elle est cultivée dans un milieu contenant de l'arséniate mais fortement carencé en phosphate (As+/P-), GFAJ-1 croit à une vitesse 40% inférieure comparativement à un milieu contenant du phosphate et pas d'arsenic (As-/P+). Cependant, dans le milieu As+/P- les bactéries ont un volume une fois et demie plus grand que dans le milieu As-/P+. Cette augmentation de volume semble être associée à l'apparition de grandes vacuoles riches en poly-β-hydroxybutyrate.
La teneur en phosphore de ces bactéries était de 0,019% (± 0,001) de leur poids sec, soit le trentième de celle trouvée lorsqu'elles sont cultivées sur un milieu ayant une concentration en phosphate de 1,5 mmol⋅L-1 et environ le centième de la teneur habituelle de la plupart des bactéries. Cette teneur en phosphore représente environ le dixième de la teneur en arsenic de ces bacilles (0,19 ± 0,25% en poids sec).
Distribution intracellulaire de l'arsenic
Pour analyser finement le devenir de l'arsenic dans la bactérie cultivée sur milieu dépourvu de phosphore, les chercheurs utilisèrent de l'arséniate radioactif. Ils observèrent qu'en phase de croissance stationnaire, 10 % de l'arséniate absorbé était incorporé dans les acides nucléiques, plus de 75 % se retrouvait dans la fraction protéique et une faible part dans les lipides[7]. Les auteurs notent tout de même que la fraction protéique obtenue par extraction au phénol comporte de nombreux composés non protéiques, et que les 75% observés sont probablement une surestimation.
Points restant à élucider et critiques
Au moment de la publication de l'article dans Science en décembre 2010, des interrogations subsistent. Il reste à déterminer si l'arsenic est effectivement incorporé dans le squelette de l'ADN, ou si l'arsenic est utilisé par la bactérie en remplacement du phosphore pour d'autres métabolismes, et si les traces infimes de phosphore toujours présent dans les milieux de culture suffisent pour fournir la quantité nécessaire à la synthèse de l'ADN[8]. Les critiques aux conclusions de l'article sur GFAJ-1 sont de trois ordres[9] :
- Dans le milieu de culture utilisé, il reste des traces de phosphore, environ 1/10000 de l'arsenic présent. Dans ces conditions, GFAJ-1 contient certes de l'arsenic (dont il n'est pas établi qu'il soit incorporé dans des molécules biologiquement actives) mais seulement trois fois plus que de phosphore. La bactérie semble donc faire tout son possible pour récupérer le peu de phosphore présent plutôt que d'utiliser l'arsenic.
- Le cytoplasme des cellules est un milieu réducteur dans lequel on s'attend à ce que les ions arseniate soient réduits en arsénite
[10]. Dans les mêmes conditions physiologiques, le phosphate n'est par contre pas réduit en phosphite. L'ADN "arsénié" ne pourrait pas exister, faute d'arseniate intracellulaire pour se substituer au rôle biologique joué par le phosphate.
- Les liaisons chimiques impliquant des atomes liés à l'arsenic (en particulier les esters d'arseniate, qui seraient nécessaires à la formation d'un ADN basé sur l'arsenic) ne durent généralement pas plus de quelques minutes en milieux aqueux, à l'inverse des molécules basées sur le phosphore qui sont beaucoup plus stables[8],[11].
La présence d'arsenic incorporé dans le squelette de l'ADN peut être démontrée par une analyse par spectrométrie de masse de l'ADN purifié. Pour Purificación Lopez, microbiologiste spécialiste des extrêmophiles au CNRS, le fait que « cette expérience ne figure pas dans le compte rendu de leurs recherches est suspect »[12]. En 2011, Rosie Redfield publie l’intégralité du protocole expérimental sur son blog et réfute les conclusions des chercheurs de la NASA[13].
Notes et références
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « GFAJ-1 » (voir la liste des auteurs)
Notes
- sept dilutions au dixième effectuées de manière successive, avec du milieu contenant 5 mmol⋅l-1 d'arséniate
Références
- Sigle de « Give Felisa A Job » (littéralement donne du travail à Felisa), Felisa Wolfe-Simon étant la première auteure de l’article relatant la découverte de ce bacille.
- Un ADN à l'arsenic : nouvelle forme de vie et révolution en exobiologie sur futura-sciences.
- http://www.futura-sciences.com/fr/news/t/biologie-3/d/bacteries-a-larsenic-non-ce-nest-pas-une-nouvelle-forme-de-vie_26485/
- La bactérie vivant d’arsenic : une exception terrestre sur maxisciences.com
- (en) « Supporting Online Material for A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus », dans Science, 2 décembre 2010 [texte intégral, lien DOI]
- (en) « A Bacterium That Can Grow by Using Arsenic Instead of Phosphorus », dans Science, 2 décembre 2010 [texte intégral, lien DOI]
- (en) Elizabeth Pennisi, « What Poison? Bacterium Uses Arsenic To Build DNA and Other Molecules », dans Sciences Magazine, 2010 [lien DOI]
- (en) Alla Katsnelson, « Arsenic-eating microbe may redefine chemistry of life », dans Nature, 2 décembre 2010 [texte intégral, lien DOI] :
« It would be good if they could demonstrate that the arsenic in the DNA is actually in the backbone. »
- (en) Simon Silver et Le T. Phung, « Novel expansion of living chemistry or just a serious mistake? », dans FEMS Microbiol Lett., vol. 315, 2011, p. 79-80 [lien PMID, lien DOI]
- (en) Barry P. Rosen et A. Abdul McDermott, « Life and death with arsenic: Arsenic life: An analysis of the recent report "A bacterium that can grow by using arsenic instead of phosphorus" », dans BioEssays, vol. 33, 2011, p. 350-357 [lien PMID, lien DOI]
- Thriving on Arsenic
- http://sciences.blog.lemonde.fr/2010/12/03/la-bacterie-qui-aime-larsenic-pourrait-reveler-des-surprises/
- (en) First evidence refuting Wolfe-Simon et al.’s results , 2 août 2011
Liens externes
- Portail de la biologie cellulaire et moléculaire
- Portail de la microbiologie
Wikimedia Foundation. 2010.