- Hélice (biochimie)
-
Les hélices sont, en biochimie, des conformations tridimensionnelles en forme d'hélice qu'adoptent certaines molécules ou certaines parties de molécules.
Sommaire
Protéines
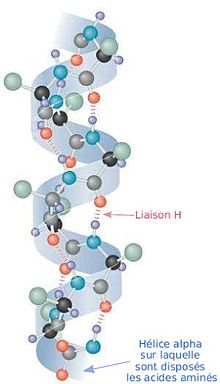
Les protéines sont des molécules organiques constituées par une certaine séquence d'acide aminés reliés entre eux par une liaison peptidique. Le groupement carbonyle (C=O) et le groupement amine (NH) de la liaison pepditique recherchent une configuration de basse énergie. L'une des manières de parvenir à minimiser l'énergie interne de la molécule, et donc de la stabiliser, consiste à créer des liaisons hydrogènes[1]. Or le groupement amine d'une liaison peptidique située en position n peut contracter une liaison de ce type avec le groupement amine d'une autre liaison pepditique située 4 acides aminés plus en aval dans la chaine. Et ainsi de suite : n+1 crée une liaison H avec n+5. Ces liaisons hydrogène forcent la protéine à adopter une conformation en hélice. Lorsque la liaison implique les acides aminés n et n+4, on parle d'hélice alpha. Lorsqu'elle concerne les acides aminés n et n+3, on parle d'hélice 310 et lorsqu'elle concerne les acides aminés n et n+5, on parle d'hélice π. On peut considérer que le feuillet béta est en fait une hélice très étirée.
L'hélice alpha permet donc, comme on vient de le voir, de diminuer l'énergie globale de la protéine. Pourquoi certaines régions de la protéine adoptent cette configuration plutôt qu'une autre demeure l'objet de recherches. Seules certaines tendances qu'ont certaines séquences d'amino acides ont été notées comme figurant plus souvent, statistiquement, dans une hélice alpha. C'est dû en grande partie au fait que les liaisons peptidiques peuvent 'choisir" d'assouvir leur liaison hydrogène avec des chaînes latérales d'acides aminés plutôt qu'avec les liaisons peptidiques.
Il existe cependant un cas où la séquence d'acide aminé doit adopter la conformation hélicoïdale : lorsqu'il s'agit de la partie d'une protéine membranaire qui traverse la membrane. En effet, dans ce milieu hydrophobe, la seule manière de réaliser des liaisons H est d'adopter une conformation en hélice (ou feuillet beta, mais nous avons vu que le feuillet béta est un cas particulier de l'hélice).
On rencontre plusieurs types d'hélices dans les protéines :
- les hélices alpha[2], dont le sens de giration est horaire. Il s'agit, de loin, de la structure en hélice la plus représentée dans les protéines. On rencontre aussi des hélices 310 droite et des hélices π. Ces trois types d'hélices sont permises par le diagramme de Ramachandran.
L'hélice alpha étant active optiquement[3], les mesures de dichroïsme circulaire permettent de déterminer le "taux d'hélicité" d'une protéine en solution[4].
Acides nucléiques
 les trois formes d'ADN : A, B, Z. On distingue sur la forme B, centrale, le petit sillon au centre de l'image, et deux parties du grand sillon en haut et en bas de l'image.
les trois formes d'ADN : A, B, Z. On distingue sur la forme B, centrale, le petit sillon au centre de l'image, et deux parties du grand sillon en haut et en bas de l'image.
Les acides nucléiques s'organisent en doubles hélices pour satisfaire les liaisons entre bases. Cette structure a pour effet de réaliser un empilement des bases les unes sur les autres, minimisant ainsi l'énergie du système en excluant l'eau. Cet empilement est d'ailleurs à l'origine de l'effet hyperchrome de l'ADN (augmentation de l'émission de fluorescence de l'ADN lorsque deux brins complémentaires se séparent). Dans une hélice d'ADN, on a donc un cœur hydrophobe constitué par l'empilement des bases les unes au-dessus des autres et un squelette constitué par les pont phosphore et les sucres (désoxyribose) plutôt hydrophile.
L'hélice de type B, la plus commune, définit un grand sillon et un petit sillon. L'accès aux bases contenues dans le grand sillon et dans le petit sillon est différent. De plus, certaines protéines de régulation se liant à l'ADN montrent un tropisme plus élevé pour l'un des sillons.
Selon les conditions du milieu (salinité, teneur en eau), mais aussi dans une certaine mesure selon la composition en bases, les acides nucléiques adoptent l'une des trois conformations suivantes :
- hélice B double hélice droite
- hélice A double hélice droite
- hélice Z double hélice gauche
Ces trois types d'hélices sont retrouvées chez l'ADN que l'on nomme alors B-ADN, A-ADN et Z-ADN. Le plus représenté est le B-ADN. L'ARN adopte l'hélice A.
Super hélice et compaction de l'ADN
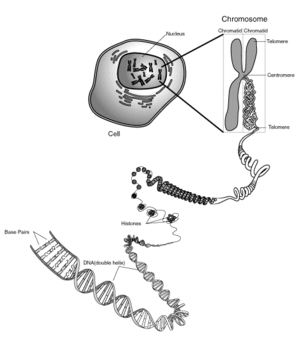
Le cas de l'hélice d'ADN est remarquable : l'hélice permet de maintenir minimale (donc stable) l'énergie interne de la molécule, mais l'hélice permet aussi la compaction de l'ADN. En effet, l'hélice d'ADN s'organise ensuite en super hélice. On peut comprendre la super hélice en observant l'hélice d'un fil de téléphone. En le torsadant, on obtient une hélice constituée d'une hélice. Chez l'homme et les organismes eucaryotes, les histones permettent un degré de compaction supplémentaire. Les six degrés de compaction permettent d'obtenir un chromosome uni et compact, indispensable pour la ségrégation des chromosomes au moment de la mitose.
Chez les procaryotes, l'ADN est de forme circulaire, c'est-à-dire qu'il est continu. Ainsi, certaines molécules comme les ADN topoisomérase[5] permettent d'augmenter ou diminuer le nombre de super enroulements[6] (enroulement en superhélice de la double hélice). Deux molécules d’ADN différant par leur nombre de spires sont nommées topoisomères. Note que si l'un des brins comporte une discontinuité (rupture d'une liaison phosphodiester du squelette), la superhélicité devient nulle. Certaines enzymes sont capables de réaliser ce "nick".
Les différents superenroulements de l'ADN permettent principalement un gain de place (une molécule d'ADN humain mesure environ un mètre et est confinée dans le noyau qui mesure environ 1 µm de diamètre) ou un accès plus facile aux protéines de transcription.
Références
- Alain Gerschel, Liaisons intermoléculaires, les forces en jeu dans la matière condensée , EDP Sciences Éditions, 1995, p. 12
- L Pauling, « The Structure of Proteins: Two Hydrogen-Bonded Helical Configurations of the Polypeptide Chain », dans Proceedings of the National Academy of Science in Washington, vol. 37, 1951, p. 205–? [lien DOI]
- Hecht, E., Optics 3rd Edition (1998) Addison Wesley Longman, Massachusetts.
- Fasman, G.D., Circular Dichroism and the Confromational Analysis of Biomolecules (1996) Plenum Press, New York.
- Champoux J, « DNA topoisomerases: structure, function, and mechanism », dans Annu Rev Biochem, vol. 70, 2001, p. 369–413 [lien PMID, lien DOI]
- Wang J, « Cellular roles of DNA topoisomerases: a molecular perspective », dans Nat Rev Mol Cell Biol, vol. 3, no 6, 2002, p. 430–40 [lien PMID, lien DOI]
Wikimedia Foundation. 2010.