- Topoisomère
-
Soient deux molécules d'ADN circulaires ayant exactement la même séquence de bases, elles peuvent cependant différer entre elles par ce que l'on appelle le nombre d'enlacements, c'est-à-dire le nombre de tours que fait l'un des brins autour de l'autre brin. On appelle alors topoisomères, ces deux ADN différant uniquement par le nombre d'enlacements.
Sommaire
Notion de tension
Le nombre d'enlacement est une constante topologique, le changement du nombre de paires de bases par tour sera donc nécessairement compensé par un surenroulement opposé. Positif ou négatif.
Différent état de topoisomères
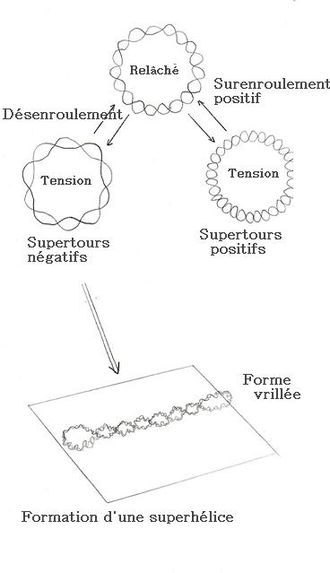
Etat relâché
La contrainte sur la double hélice est minimale, c'est la configuration la plus stable. L'ADN en forme B comporte 10 paires de bases par tours. Dans les cellules, l'ADN a une configuration proche de celle-ci, avec en moyenne 10,5 paires de bases par tour au lieu de 10. Cette valeur peut varier en fonction des différentes protéines associées à l'ADN.
Etat surenroulé
L'axe de la double hélice peut s'enrouler sur lui-même pour former une superhélice, deux formes possibles.
Surenroulement positif
Le nombre d'enlacement a été augmenté. Formation d'une superhélice gauche.
Surenroulement négatif
Le nombre d'enlacement a été diminué. Formation d'une super hélice droite.
Surenroulement négatif=désenroulement.
Le surenroulement négatif est le plus rencontré dans la nature car il permet une réduction de la taille globale de la molécule d'ADN. Le désenroulement le plus courant est de un supertour pour 200 paires de bases. En effet, le surenroulement négatif provoque une contrainte qui induit un enroulement de l'axe de l'hélice d'ADN, qui se vrille alors. Il présente alors deux avantages :
- la formation de la superhélice rend plus compacte la molécule, notamment dans les bactéries ou les mitochondries où l'ADN est circulaire ;
- il permet d'absorber une partie de la contrainte générée par le déroulement local nécessaire à la réplication et à la transcription dans un ADN circulaire. En effet, lorsqu'une ADN polymérase ou une ARN polymérase utilise un ADN circulaire comme matrice, ces enzymes doivent créer une zone simple brin en déroulant la double-hélice.
Le surenroulement négatif est créé de manière active chez les bactéries par une enzyme appelée ADN gyrase qui appartient à la famille des topoisomérases II.
- Portail de la chimie
- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.