- Polymorphisme nucléotidique
-
 Pour les articles homonymes, voir SNP.
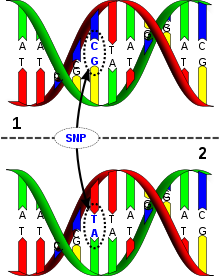
Pour les articles homonymes, voir SNP.Le polymorphisme nucléotidique ou polymorphisme d'un seul nucléotide (SNP, single-nucleotide polymorphism) est, en génétique, la variation (polymorphisme) d'une seule paire de bases du génome, entre individus d'une même espèce. Ces variations sont très fréquentes (environ une paire de bases sur mille dans le génome humain[1],[2]).
Les SNP représentent 90 % de l'ensemble des variations génétiques humaines, et des SNP avec une fréquence allélique supérieure à un pour cent sont présents toutes les cent à trois cent paires de bases en moyenne dans le génome humain, où deux SNP sur trois substituent la cytosine avec la thymine.
Sommaire
Localisation
Les SNP peuvent se retrouver au sein de régions codantes de gènes (exon), de régions non codantes de gènes (intron), ou de régions intergéniques, entre les gènes. Dans le cas où les SNP se retrouvent au sein des régions codantes, celles-ci ne vont pas obligatoirement modifier la séquence d'acide aminé de la protéine produite, et ce, grâce à la redondance du code génétique.
Les SNP qui se retrouvent dans des régions non codantes peuvent avoir des conséquences sur l'épissage, les facteurs de transcription, ou sur les séquences d'ARN non codant.
Types
On parlera de formes alléliques synonymes dans le cas où plusieurs formes d'un SNP mènent à la même séquence polypeptidique, et de formes non synonymes dans le cas où les séquences produites diffèrent.
Utilisation
Les SNP sont des outils permettant d'identifier des génotypes (reconnaître des personnes par exemple), à partir d'échantillons de matière organique, ou permettant de contribuer à la construction d'arbres généalogiques d'êtres vivants ou d'espèces.
Notes et références
- Neil Campbell et Jane Reece, Biologie, 7ème édition, Pearson Education, 2007, p. 438.
- http://www.futura-sciences.com/fr/doc/t/genetique/d/diversite-genetique-et-question-de-races_786/c3/221/p6/
Lien externe
- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.