- G-quadruplexe
-
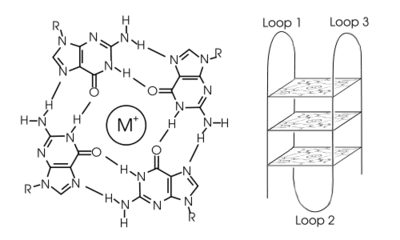
Les G-quadruplexes(G4) sont des structures secondaires à quatre brins que peuvent adopter les acides nucléiques (ADN ou ARN) riches en guanines. Cette structure repose sur des appariements de base de type Hoogsteen formant un plateau de 4 guanines (G), aussi appelé « quartet ». L’empilement parallèle et ininterrompu d’au moins 2 quartets, intercalés par un cation monovalent (sodium ou potassium) stabilisant la structure, constitue le G4. Ceci implique une structure primaire d’ADN contenant 4 paires de G pouvant se situer sur la même molécule d’ADN (G4 intramoléculaire) ou sur des molécules d’ADN différentes (G4 intermoléculaire).
Sommaire
Découverte et caractérisation
Les G-quartets et les structures G-quadruplexes ont été caractérisées pour la première fois en 1962 par diffraction de rayon X[1]. Elles ont été étudiées de manière détaillée in vitro, et il a été montré qu’elles se formaient dans des conditions de salinité et de pH physiologiques[2], et que de nombreuses protéines étaient capables de fixer, stabiliser, ou au contraire dérouler ces structures in vitro (pour revue[3]).
Les G-quadruplexes existent dans le vivant
Des études bioinformatiques récentes révèlent une forte présence de séquences potentiellement capables de former des G-quadruplexes dans tous les génomes analysés jusqu’alors. Ces séquences sont fortement enrichies à certains loci notamment au niveau des promoteurs, et cela concerne plus particulièrement certaines classes de gènes, alors que d’autres en sont dépourvus. Ceci suggère un rôle conservé de contrôle transcriptionnel, d’épissage ou de traduction pour ces structures secondaires. Outre ce rôle putatif dans le contrôle de l’expression des gènes, les G4 semblent intervenir dans de nombreux autres processus cellulaires, comme la biogénèse des ribosomes et la maturation des ARN ribosomiques, la recombinaison homologue, la régulation de la taille des télomères par inhibition de la télomérase, et l’inhibition de la réplication des ADN ribosomiques et des télomères. L’implication des G4 dans ces mécanismes où l’ADN est activement ouvert est cohérente avec l’idée que le G4 ne peut se former que lorsque l’ADN (ou l’ARN) se trouve sous forme simple brin. Plusieurs études ont récemment mis en évidence l'existence des G-quadruplexes dans les organismes vivant. Chez les ciliés par visualisation directe à l’aide d’anticorps extrêmement affins des G-quadruplexes formés par les télomères de Stylonychia lemnae [4] ainsi que le rôle de deux protéines télomériques (TEBPα et ß) dans la formation de cette structure démontrant un rôle important des G-quadruplexes dans le métabolisme des télomères[5]. Chez la bactérie, la formation de G-quadruplexes au cours de la transcription a été mis en évidence par microscopie électronique chez E. coli[6].
 Structure 3D d'un G-quadruplexe formé par la séquence télomérique humaine (brin G) (PDB ID 2HY9). Le squelette phosphate est représenté par un tube. Le centre de la structure contient 3 empilements successifs de G-quartet. Les liaisons hydrogènes sont représentées par des lignes bleues.
Structure 3D d'un G-quadruplexe formé par la séquence télomérique humaine (brin G) (PDB ID 2HY9). Le squelette phosphate est représenté par un tube. Le centre de la structure contient 3 empilements successifs de G-quartet. Les liaisons hydrogènes sont représentées par des lignes bleues.Références
- GELLERT M, LIPSETT MN, DAVIES DR. Helix formation by guanylic acid.Proc Natl Acad Sci U S A. 1962 Dec 15;48:2013-8
- Parkinson GN, Lee MP, Neidle S. Crystal structure of parallel quadruplexes from human telomeric DNA. Nature. 2002 Jun 20;417(6891):876-80. Epub 2002 May 26.
- In vivo veritas: Using yeast to probe the biological functions of G-quadruplexes. Johnson JE, Smith JS, Kozak ML, Johnson FB. Biochimie. 2008 Feb 21
- Schaffitzel C, Berger I, Postberg J, Hanes J, Lipps HJ, Plückthun A. In vitro generated antibodies specific for telomeric guanine-quadruplex DNA react with Stylonychia lemnae macronuclei. Proc Natl Acad Sci U S A. 2001 Jul 17;98(15):8572-7
- Paeschke K, Simonsson T, Postberg J, Rhodes D, Lipps HJ. Telomere end-binding proteins control the formation of G-quadruplex DNA structures in vivo. Nat Struct Mol Biol. 2005 Oct;12(10):847-54
- Duquette ML, Handa P, Vincent JA, Taylor AF, Maizels N.Intracellular transcription of G-rich DNAs induces formation of G-loops, novel structures containing G4 DNA.Genes Dev. 2004 Jul 1;18(13):1618-29.
Liens externes
Livres
Sites internet dédiés aux G-quadruplexes
- Quadruplex.org - a website to serve the quadruplex community
- Quadbase - downloadable data on predicted G-quadruplexes
- Greglist - a database listing potential G-quadruplex regulated genes
- Database on Quadruplex information: QuadBase from IGIB
- GRSDB- a database of G-quadruplexes near RNA processing sites.
- GRS_UTRdb- a database of G-quadruplexes in the UTRs.
- G-quadruplex Resource Site
Outils de prédiction de G-quadruplexes
- Quadparser: Downloadable program for finding putative quadruplex-forming sequences
- QGRS Mapper: a web-based application for predicting G-quadruplexes in nucleotide sequences and NCBI genes
- Quadfinder: Tool for Prediction and Analysis of G Quadruplex Motifs in DNA/RNA Sequences from IGIB
- Portail de la biologie cellulaire et moléculaire
- Portail de la biochimie
Wikimedia Foundation. 2010.