- Traduction de l'ADN
-
Traduction génétique
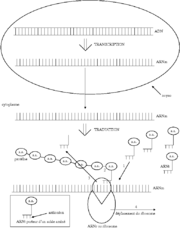
La traduction est l'interprétation des codons de l'ARNm en acides aminés. Le code génétique est le système de correspondances (code) permettant à l'ARN d'être traduit en protéine par une cellule. La traduction s'effectue dans le cytoplasme, elle nécessite des acides aminés qui sont polymérisés selon l'ordre donné par les codons de l'ARNm. Elle a aussi besoin d'énergie, des ribosomes, des ARNt, de l'activité enzymatique et d'un plan apporté par l'ARNm.
Sommaire
Processus
On peut diviser la traduction en trois phases principales : l'initiation, l'élongation et la terminaison.
L'initiation
Une sous-unité du ribosome se fixe sur l'extrémité de l'ARNm, accompagnée de l'ARNt qui porte la méthionine (dont l'anticodon est UAC). Le ribosome et la sous-unité se déplacent jusqu'à ce qu'ils parviennent au codon AUG, codant la méthionine : ce codon est le codon initiateur. Une autre sous-unité ribosomiale vient alors se poser sur ce codon. La synthèse commence.
Pour plus de détails : Les sous-unités du ribosome possèdent 3 sites : a(aminoacylARNt), p(peptidoacylARNt), e(exit). Divers facteurs protéiques rentrent en jeu. Nous allons nous intéresser au cas des eucaryotes tout d'abord.
Il existe 3 types d'initiations: l'initiation par balayage, IRES et shunt. (cf. liens externes)
L'initiation par balayage : eIF 3 se fixe dans e de la petite sous-unité et eIF 1A dans a. Ceci empêche la fixation de la grande sous-unité sur la petite. Puis eIF 5B se fixe sur 1A et eIF 2 sur 5B. 5B et 2 sont liés à du GTP et eIF 2 est également lié à l'ARNt initiateur (méthionine). On a formé le complexe 45S. Au niveau de l'ARNm en 5' on trouve eIF 4F composé de 4E 4G etA. B est également là pour éliminer les structures secondaires de l'ARN. Il y a interaction entre 4F et 3 créant ainsi une sorte de boucle au niveau de l'ARN. Cette boucle se déplace, c'est le mécanisme de scanning et quand l'ARNt initiateur en p, chargé de méthionine, est en face du codon initiateur il existe diverses modifications. Le GTP d'eIF 2 est hydrolysé en GDP, ce qui entraîne le départ d'eIF2, suivi de celui de 3 de 4B de 5B-GDP et d'eIF1A. La grande sous-unité peut alors se fixer et l'élongation peut démarrer.
L'élongation
Le ribosome se déplace alors de codon en codon au niveau de l'ARNm, et associe chaque codon à un ARNt lui correspondant qui apporte le bon acide aminé au bon endroit. Un cycle d'élongation permet d'accrocher un acide aminé. Ce nouvel acide aminé est relié au peptide en cours d'élongation grâce à une liaison peptidique créée par un ribozyme qui est ici l'ARNr 28S contenu dans la grande sous-unité ribosomique. L'énergie nécessaire à la formation de cette liaison peptidique est contenue dans la liaison ARNt - acide aminé de sorte que la formation des liaisons peptidiques ne nécessite aucune énergie extérieure (pas de consommation d'ATP). Les ARNt déchargés de leur acide aminé sont libérés au fur et à mesure. La chaîne d'acides aminés s'allonge suivant un ordre précis donné par les codons de l'ARNm.
La terminaison
Quand le ribosome parvient au niveau d'un codon stop (il en existe 3 dans tout le code génétique : UGA, UAG ou UAA, ne correspondant à aucun acide aminé) il y a action des facteurs de terminaison. Ces facteurs sont au nombre de deux chez les eucaryotes (eRF1 et eRF3) et au nombre de 3 chez les procaryotes (RF1, RF2, RF3). Les facteurs de classe I (eRF1 et RF1, RF2) reconnaissent directement les codons stop, alors que les facteurs de classe II ont une activité GTPase qui a pour but de stimuler le relargage des facteurs de classe I. On peut noter ici une différence majeure, qui est que le facteur eucaryote eRF1 reconnaît les trois codons stop, alors que chez les procaryotes RF1 reconnaît les codons UAG et UAA, et RF2 UGA, UAA. Ce dernier facteur est exprimé grâce à un événement de frameshift qui sert de mécanisme d'autorégulation traductionnelle du facteur RF2. Chez les eucaryotes le mécanisme de dissociation et de recyclage du ribosome demeurent largement incompris alors qu'il est beaucoup mieux décrit chez les procaryotes. Chez ces derniers un quatrième facteur (le RRF) agit en combinaison avec les facteurs IF3 et EFG pour provoquer la dissociation complète des deux sous unités du ribosome. L'action de ces facteurs a pu être visualisée par cryo-EM.
Note
Un ARNm peut être traduit par plusieurs ribosomes à la fois. L'ensemble formé par un ARNm et plusieurs ribosomes se déplaçant dessus s'appelle un polysome. Comme le phénomène de traduction a lieu plusieurs fois en même temps, il est donc fréquent de trouver des protéines identiques fabriquées à partir d'un même ARNm. On dit que la traduction est amplifiée.
Lien interne
Liens externes
- Baptiste Deleplace (2003), La traduction (une approche ludique de la traduction de l'ARN m en chaîne polypeptidique).
Voir aussi
- Portail de la biologie cellulaire et moléculaire
- Portail de la biochimie
Catégories : Expression génétique | Génétique | Biologie cellulaire | Biochimie
Wikimedia Foundation. 2010.