- Polymerase Chain Reaction
-
Réaction en chaîne par polymérase
 Pour les articles homonymes, voir PCR.
Pour les articles homonymes, voir PCR.La réaction en chaîne par polymérase (PCR est l'abréviation anglaise de polymerase chain reaction, l'acronyme français ACP pour Amplification en Chaîne par Polymérisation est très rarement utilisée), est une méthode de biologie moléculaire d'amplification génique in vitro, qui permet de copier en grand nombre (avec un facteur de multiplication de l'ordre du milliard), une séquence d'ADN ou d'ARN connue, à partir d'une faible quantité (de l'ordre de quelques picogrammes) d'acide nucléique (séquence spécifique d’ADN (l’Amplicon) ou amorces spécifiques constituées d'oligonucléotides de synthèse de 20 à 25 nucléotides). Cette technique permet entre autres de détecter la présence du virus VIH ou de mesurer la charge virale (concentration du virus dans le plasma), des OGM (organismes génétiquement modifiés), des virus des hépatites B, C et D. De plus en plus utilisée en criminalistique, cette technique est basée sur la combinaison de deux facteurs :
- Les propriétés de synthèse enzymatique et d’initiation « ADN double brins spécifique » des « ADN polymérases ADN dépendantes thermostables ».
- Les propriétés d’hybridation et de deshybridation des brins complémentaires d’ADN en fonction de la température.
Ces éléments permettent de contrôler l’activité enzymatique grâce à des transitions de température (assurées par un thermocycleur) répétées de manière cyclique (cf. réaction en chaîne).
Les premières ADN polymérases utilisées provenaient d'une bactérie thermophile (résistante à des températures très élevées), par exemple Thermus aquaticus (Taq polymérase) ou encore Pyrococcus furiosus (Pfu polymérase), Thermococcus litoralis (Vent ou Tli polymérase), Thermus thermophilus (Tth polymérase). De nos jours, les enzymes utilisées sont dites recombinantes, ce qui simplifie considérablement leur obtention, et leurs propriétés ont été largement modifiées pour les rendre plus efficaces, plus fidèles…
En moins de dix ans, cette technique (maintenant capable de faire plus d'un milliard de copies en moins d'une heure) s'est imposée dans les laboratoires et a révolutionné la biologie moléculaire.
Historique
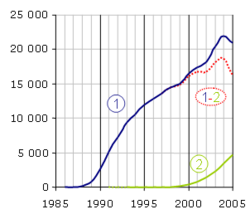
La PCR est une technologie qui a bouleversé la biologie moléculaire et s'est implantée très rapidement dans les laboratoires. En revanche, la PCR en temps réel a dû attendre la mise sur le marché d'un certain nombre d'innovations technologiques avant de se développer et est encore considérée comme une méthodologie nouvelle. Le nombre d'articles par année répondant aux mots clés (1) "polymerase chain reaction" et (2) "real-time polymerase chain reaction" sur le moteur de recherche PubMed donne une assez bonne idée de leur importance dans le monde scientifique. Notez que la méthode n'est pas exempte de biais, par exemple quelques articles sont trouvés pour la PCR en temps réel en 1991 et 1992 (en pointillé) alors que son principe n'a été décrit qu'en 1993. La différence (1-2) est représentative du poids de la PCR en point final, qui va probablement céder progressivement la place au temps réel.
Cette technique a largement évolué depuis ses débuts. Parmi les évolutions les plus fondamentales, on retrouve :
- Le remplacement des fragments de Klenow d’ADN polymérase I d’E . coli par une polymérase thermorésistante (initialement la Taq) qui évite de devoir remettre de l’enzyme à chaque cycle. Cette innovation permet un bond énorme vers l’automatisation et évite de devoir ouvrir le tube réactionnel, limitant considérablement le risque de contamination.
- La généralisation des thermocycleurs (un bon nombre d’anciennes expérimentations ont été réalisées avec trois bains-marie) a permis de rendre la PCR moins contraignante, plus reproductible et était un pré requis indispensable à la plupart des applications nouvelles.
- L’invention de la PCR en temps réel qui permet de rendre la méthode quantitative et évite plusieurs étapes expérimentales contraignantes, telles l’électrophorèse sur gel d’agarose, l’acquisition de fluorescence, la calibration de l’acquisition du signal, etc.
Par souci de clarté, les dates correspondent à la première publication sur le domaine et seuls les premiers auteurs sont cités, les références complètes étant dans le chapitre "bibliographie". Ce choix permet en outre de limiter les polémiques telles le rôle de Rosalind Elsie Franklin dans la découverte de la structure de la double hélice d’ADN .
- 1953 : Découverte de la structure en double hélice de l’ADN par James Dewey Watson et Francis Harry Compton Crick, (prix Nobel de physiologie ou médecine en 1962).
- 1956 : Découverte de l’ADN polymérase ADN dépendante (ADN pol I) par Arthur Kornberg (prix Nobel de physiologie ou médecine en 1959).
- 1970 : Co-découverte indépendante de l’ADN polymérase ARN dépendante par Temin HM et Baltimore D (prix Nobel de physiologie ou médecine en 1975).
- 1986 : Première publication publique sur la PCR par Kary Mullis (prix Nobel de chimie en 1993).
- 1988 : Première PCR réalisée avec une ADN polymérase thermostable, provenant de Thermus aquaticus, par Saiki RK.
- 1991 : Première détection du produit de PCR par sonde (sonde d’hydrolyse) par Holland PM.
- 1992 : Invention de la PCR en temps réel par Higuchi R.
- 1995 : Première publication sur la TAIL-PCR par Liu YG
- 1996 : Mise au point des polymérases temporairement inactives et activables par la chaleur par Birch DE.
- 1997 : Mise en évidence de la variation de température "Tm dépendante" du SYBR green par Wittwer CT.
- 1997 : Première discrimination d’allèle grâce à la courbe de fusion par Lay MJ.
Principe
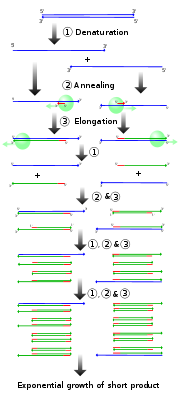
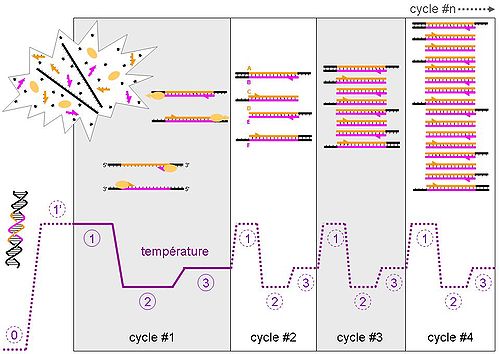
La PCR est une technique basée sur une répétition de cycles de transition de température. Sauf pour certaines méthodologies (par exemple l’utilisation de sondes d’hydrolyse), chaque cycle contient trois étapes détaillées ci-dessous. Par souci de didactisme, nous allons considérer pour l’exemple qui suit une efficacité de PCR de 100%.
Les différentes étapes de la PCR
Conditions natives (0 sur le schéma)
Cette étape se fait généralement à température ambiante. L’ADN bicaténaire adopte sa conformation en double hélice. Dans cet exemple, nous considérerons qu’il n’y a qu’une molécule initiale d’ADN double brin dans la solution, la zone colorée (rose et orange) correspondant à notre amplicon. Note: les ADN complémentaires issus de la transcription inverse sont généralement simple brin et adopteront alors de complexes conformations tridimensionnelles similaires à celle des ARN.
Dénaturation initiale (1’ sur le schéma)
Avant de commencer les cycles de PCR proprement dit, une étape de chauffage (généralement 10 à 15 minutes à 95°C) est réalisée. Cette étape permet de : déshybrider les ADN double brin, de casser les structures secondaires, d’homogénéiser le milieu réactionnel par agitation thermique, d’activer les polymérases de type « Hot start », de dénaturer d’autres enzymes qui pourraient être dans la solution (Transcriptase Inverse, Uracil-N-Glycosylase).
Phase de dénaturation (1 sur le schéma)
Cette étape (généralement 0 à 1 minute à 95°C) permet de déshybrider les ADN, de « décrocher » les polymérases qui seraient encore liées à une matrice et d’homogénéiser le milieu réactionnel.
Phase d’hybridation ou d'appariement des amorces (2 sur le schéma)
Cette étape (généralement 2 à 60 secondes à 56-64°C) permet aux amorces sens et anti-sens de s’hybrider aux ADN matrice grâce à une température qui leur est thermodynamiquement favorable. Peu de brins d’ADN matrice peuvent s’hybrider (se lier) avec leur brin complémentaire, ce qui empêcherait la fixation des amorces, car ces dernières sont bien plus courtes et en concentration bien plus importante. Expérimentalement, il est constaté que la PCR fonctionne même avec une phase d’hybridation avec une température supérieure de quelques degrés au Tm théorique des amorces, probablement parce qu’elles interagissent déjà avec les polymérases, qui stabiliseraient leur hybridation à l’ADN matrice.
Phase d’élongation (3 sur le schéma)
Cette étape (généralement 4 à 120 secondes à 72°C) permet aux polymérases de synthétiser le brin complémentaire de leur ADN matrice à une température qui leur est optimale. Ce brin est fabriqué à partir des dNTPs libres présents dans le milieu réactionnel. La durée de cette étape dépend normalement de la longueur de l’amplicon.
Evolution de l'ADN au cours des 4 premiers cycles
Cycle n°1
-
- Lors de la phase 1, nous constatons que l’ADN initial a adopté une conformation « linéaire » (sans structure secondaire) et simple brin. Les amorces, les dNTPs et les polymérases sont en large excès et répartis de façon homogène dans la solution.
- Lors de la phase 2, une des amorces sens s’hybride avec sa séquence complémentaire sur le brin anti-sens (en rose), une des amorces anti-sens se liant elle au brin sens (en orange). Deux polymérases peuvent alors interagir avec les deux complexes amorces/ADN matrice.
- Lors de la phase 3, les polymérases parcourent leur brin matrice de son extrémité 3’ vers son extrémité 5’ tout en synthétisant le brin complémentaire. Elles s’arrêteront à la fin du cycle, décrochées par la phase de dénaturation du cycle suivant. Les ADN néo-synthétisés sont donc précisément définis à leur extrémité 5’ mais pas à leur extrémités 3’ (parties noires). Les ADN sont alors bicaténaires sur une longueur plus ou moins importante.
- A la fin de l’étape 3, nous avons alors deux brins d’ADN matrice et deux brins (un sens et un anti-sens) d’ADN précisément définis à leur extrémité 5’ uniquement.
Cycle n°2
Les trois phases se déroulent de la même manière qu’au cycle n°1, sauf que deux polymérases arrivées au bout de leur ADN matrice se décrochent spontanément. À la fin de la phase 3, nous obtenons tous les types d’ADN qui existeront lors de la PCR, soit :
-
- Un brin d’ADN natif sens (A).
- Deux brins d’ADN sens précisément définis à leur extrémité 5’ uniquement (B).
- Un brin d’ADN anti-sens correspondant à l’amplicon, c'est-à-dire précisément défini à ses deux extrémités (C).
- Deux brins d’ADN anti-sens précisément définis à leur extrémité 5’ uniquement (D).
- Un brin d’ADN sens correspondant à l’amplicon, c'est-à-dire précisément défini à ses deux extrémités (E).
- Un brin d’ADN natif anti-sens (F).
Cycle n°3
Idem au cycle 2. À la fin de la phase 3, nous observons 1 brin de type A et F, 3 de B et D, et 4 de C et E. Nous observons l’apparition de deux molécules d’ADN double brins C-E qui correspond à notre amplicon.
Cycle n°4
Idem au cycle 3. À la fin de la phase 4, nous observons 1 brin de type A et F, 4 de B et D, et 11 de C et E. Nous observons que l’amplicon devient la combinaison majoritaire.
Cycles au-delà de 4
Si nous avions augmenté le nombre de cycles, nous aurions obtenu le tableau suivant :
nombre de molécule de chaque type en fonction du cycle de PCR cycle numéro : 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 n type de molécule A 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 = An − 1 + 0 B 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 = Bn − 1 + 1 C 0 0 1 4 11 26 57 120 247 502 1013 2036 4083 8178 16369 32752 = Cn − 1 + (Bn − 1 + En − 1) D 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 = Dn − 1 + 1 E 0 0 1 4 11 26 57 120 247 502 1013 2036 4083 8178 16369 32752 = En − 1 + (Cn − 1 + Dn − 1) F 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 = Fn − 1 + 0 simple brin nombre 2 4 8 16 32 64 128 256 512 1024 2048 4096 8192 16384 32768 65536 = Σ An à Fn % amplicon 0,00 0,00 25,00 50,00 68,75 81,25 89,06 93,75 96,48 98,05 98,93 99,41 99,68 99,83 99,91 99,95 = 100 * (Cn + En) / Σsimplebrin double brins nombre 1 2 4 8 16 32 64 128 256 512 1024 2048 4096 8192 16384 32768 = Σsimplebrin / 2 % amplicon 0,00 0,00 0,00 12,50 43,75 65,63 79,69 88,28 93,36 96,29 97,95 98,88 99,39 99,67 99,82 99,91 = 100 * ((Cn − Bn) + (En − Dn)) / Σdoublebrin
En analysant ce tableau, nous constatons que :- Les molécules d’ADN natif (A et F) ne sont pas dupliquées.
- Les molécules précisément définies à leur extrémité 5’ uniquement (B et D) augmentent de manière linéaire (de 1 par cycle sauf si l’ADN natif se dégrade).
- Le nombre de molécules simple ou double brin augmente selon une exponentielle d’ordre 2.
- Les molécules contenant la séquence exacte à amplifier (C et E) apparaissent dès le deuxième cycle et augmentent selon une suite arithmétique qui tend vers une exponentielle d’ordre 2 lorsque le nombre de cycles augmente. Notez que dès le dixième cycle, elles représentent près de 99% de l’ensemble.
- L’amplicon (les couples C-E) apparaît dès le troisième cycle et augmente selon une loi de même type que ses composants. Il représente près de 98 % des molécules au dixième cycle, 99,91 % au quinzième (zone où les ADNc issus d’ARNm fortement exprimés commencent généralement à devenir détectables).
Ces valeurs ont été obtenues en partant d’une seule molécule initiale d’ADN double brins, mais autrement, chaque matrice aurait subi le même processus. À un cycle donné, la quantité d’ADN dépend donc du nombre initial de matrices. En revanche, quelle que soit sa concentration initiale, il est théoriquement possible d’obtenir n’importe quelle quantité en ajustant le nombre de cycles. La PCR est donc régie théoriquement par la loi :
[ADN]cyclen = [ADN]initialexEn
Mais la PCR est une réaction enzymatique complexe. Le produit est identique au substrat et peut venir inhiber l’enzyme, mais surtout, les réactifs secondaires (amorces, dNTP) peuvent commencer à manquer. La PCR ne peut donc avoir une loi d’amplification exponentielle que tant que l’ADN matrice est le seul facteur limitant. La réaction devient ensuite imprédictible et il est alors impossible de pouvoir comparer plusieurs échantillons entre eux sans un biais quantitatif plus ou moins significatif (voir l’article sur la PCR quantitative). Il est donc important de bien comprendre les différentes phases d’une cinétique de PCR.
Cinétique mesurable d'une PCR
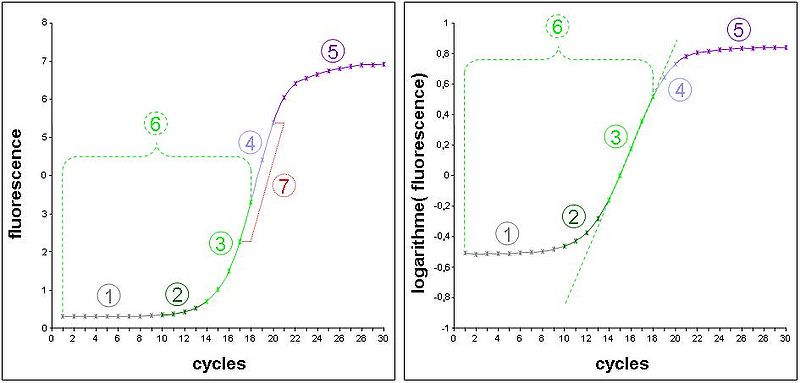
Ce chapitre traite des cinétiques mesurables d'une PCR, car des limitations technologiques rendent inaccessibles les cycles précoces, et généralement une large portion de la partie exponentielle. Son profil apparent (mesurable) adopte plusieurs phases distinctes, plus ou moins développées en fonction de choix méthodologiques.
- Schéma de gauche : données expérimentales d’une cinétique de PCR mesurée sur un thermocycleur en temps réel. La fluorescence émise par un intercalant de l’ADN est proportionnelle à la quantité d’ADN présent (cad majoritairement l’amplicon). La courbe représente donc la cinétique d’amplification de l’ADN cycle par cycle.
- Schéma de droite : les valeurs de fluorescence précédentes ont été converties en logarithme décimal. Une cinétique de type exponentiel devrait donc être transformée en un segment de droite (segment quantifiable).
- 1) Bruit de fond de la fluorescence aspécifique du marqueur. Son niveau est très dépendant de la nature du marqueur et du soluté de l’échantillon.
- 2) Zone où la mesure de fluorescence est biaisée par le bruit de fond. Elle se distingue aisément sur le schéma de droite car elle adopte un profil d’exponentielle, alors que les valeurs sont déjà dans un repère semi-logarithmique.
- 3) Zone de la cinétique en phase exponentielle et mesurable sans biais. Elle adopte dans le schéma de gauche l’apparence d’un segment de droite nommé segment quantifiable. Remarquez que dans cet exemple expérimental, il ne fait que 5 cycles.
- 4) Zone de la cinétique où la polymérase devient un facteur limitant. L’accumulation d’amplicon se fait alors de manière constante et devient linéaire sur le schéma de gauche.
- 5) Phases d’amortissement puis de plateau de la cinétique dues à l’apparition de facteurs limitants non constants (dNTP, fluorophore, etc.), de dégradation de l’activité enzymatique, de la qualité de fluorescence…
- 6) Phase de la cinétique qui répondait réellement à une loi exponentielle, mais seule la zone 3 est réellement exploitable (sauf pour le maxima de dérivé seconde en PCR en temps réel).
- 7) Zone linéaire sur le schéma de gauche. Une erreur classique en PCR en point final était de déterminer cette zone grâce à une gamme de cycles pour tenter de faire une mesure quantitative de l’ADNc initial à ce niveau.
Efficacité de la PCR
L’avènement de la PCR en temps réel a mis en exergue que l’efficacité d’une réaction de PCR (généralement notée E) n’était pas toujours égale à deux. Cela signifie que sur l’ensemble des cycles considérés comme quantitatif (zone n° 6 de la cinétique), tous les brins matrices ne servent pas forcément à donner une copie complète de l’amplicon. Deux phénomènes en sont les principales causes :
- Tous les brins matrices ne sont pas forcément liés par un complexe amorce/polymérase lors de la phase d’hybridation. La probabilité d’amorçage peut être influencée par la température, la longueur et la séquence de l’amorce, sa composition en nucléotides naturels ou modifiés, sa concentration dans la solution, la composition ionique de cette dernière, l’auto-hybridation entre les amorces ou la compétition possible d’ADN non cible (par exemple un pseudo-gène).
- Toutes les synthèses ne sont pas forcément complètes, notamment si la phase d’élongation est trop courte. Un brin incomplet à son extrémité 3’ ne peut pas servir de matrice car l’amorce complémentaire ne peut pas se lier à lui.
Cette efficacité est donc théoriquement inférieure ou égale à deux mais certaines sources considèrent qu’elle est acceptable jusqu’à 2,3!
Ce qui suit peut être un élément de controverse !
L’auteur de ce chapitre n’a pour l’instant trouvé aucune source justifiant cette position. Il considère qu’il y a deux types d’explication possible :
- Une part significative (jusqu’à 30%) des brins d’ADN servent plusieurs fois de matrice pendant un cycle et cette proportion est conservée pendant toute la phase exponentielle. Donc pour chacune de ces matrices, un complexe amorce/polymérase s’est fixé une première fois, une synthèse complète a été effectuée, l’ADN double brin s’est déshybridé à une température extrêmement défavorable, un second complexe amorce/polymérase s’est fixé à une température probablement défavorable (étape d’élongation) et qu’une seconde synthèse complète a eu lieu.
- Un biais expérimental (dans un facteur de dilution par exemple) ou d’analyse mathématique (généralement commis par l’utilisation inappropriée d’outil d’optimisation présent dans le logiciel du thermocycleur) a été commis. Ces deux types de biais ont été plusieurs fois constatés.
La majorité des protocoles expérimentaux donne une efficacité de PCR entre 1,75 et 2. Deux écoles s’affrontent alors, avec des arguments expérimentaux à l’appui. L’une considère que cette efficacité est une constante pour chaque amplicon dans un protocole expérimental donné. L’autre estime qu’elle varie toujours significativement et qu’elle nécessite d’être constamment remesurée. Il convient de noter qu’il est très difficile de savoir si une variation d’une efficacité de PCR observée vient de la nature même de cette méthode, d’une variation dans le protocole expérimental (manque de reproductibilité des réactifs ou de la manipulation) ou d’une variation dans l’acquisition des données (variations de fluorescence, canaux de lecture différents, biais dans l’analyse mathématique). Il est également très ardu d’être certain que l’efficacité utilisée (mesurée à chaque expérience ou non) est bien celle qui a eu cours dans l’échantillon à calibrer (voir la PCR quantitative.
Le terme efficacité peut aussi avoir deux significations selon les auteurs :
- Les premiers désignent l’ordre de l’exponentielle. L’équation de la cinétique s’écrit donc :
[ADN]n = [ADN]initialexEn
- Les seconds désignent la fraction de molécule d’ADN servant effectivement de matrice. L’équation devient alors :
[ADN]n = [ADN]initialex(1 + E)n
La deuxième formule facilite l’expression en pourcentage (en multipliant E par 100), mais que les deux concepts sont absolument identiques.
Cette efficacité de PCR est un élément fondamental à prendre en compte pour obtenir une mesure quantitative ou établir un protocole de PCR multiplexe, mais elle est généralement négligeable pour un résultat qualitatif ou en PCR en point final.
Techniques associées à la PCR
Les sigles et les noms en anglais sont donnés entre parenthèses.
PCR multiplexe
La PCR multiplexe (multiplex PCR) est un protocole destiné à amplifier plus d’un amplicon à la fois, par l'utilisation d'au moins trois amorces par réaction de PCR. Les produits de PCR ne seront alors compétitifs que pour la polymérase, les dNTP et, éventuellement, le marqueur d’ADN. Il est également possible d’amplifier différents types d’ADN reconnus par un même couple d’amorces, tels les mimics. La PCR multiplexe peut se faire en point final (les produits de PCR étant usuellement différenciés par leur taille ou la présence d’un site de restriction) ou en temps réel (chaque produit étant mesuré par une sonde spécifique couplée à un fluorophore dont le spectre d’émission est différent des autres). Ses applications qualitatives sont nombreuses (détection de souche virale, de mutations, …) mais son aspect quantitatif ne fait pas l’unanimité, malgré de fortes pressions industrielles pour ce marché très lucratif.
Méta-PCR
Meta-PCR est une méthode qui permet de donner une molécule d'ADN synthétique comprenant n'importe quelle combinaison d'ADN amplifiable par PCR dans n'importe quel ordre. Elle est effectuée dans un seul tube et se compose de deux différentes étapes de la PCR séparées par un cycle de dégel pour élimminer l'activité résiduelle de la polymérase. La Meta-PCR peut être utilisée pour accroître le débit des méthodes de numérisation et balayage des mutations .
PCR emboîtée
La PCR emboîtée, PCR gigogne ou PCR nichée (Nested PCR) est une PCR en deux étapes successives, avec deux couples d’amorce différents, le second liant des séquences situées à l’intérieur du premier amplicon. Cette technique était initialement utilisée pour réduire le risque de contamination (le produit final devait pouvoir interagir avec deux couples d’amorces, donc deux niveaux de spécificité). Elle est maintenant très utilisée par les virologues travaillant sur les virus à ARN qui peuvent avoir une haute mutabilité. Le premier couple d’amorces est conçu pour pouvoir accrocher les quelques parties stables du génome viral, le deuxième pour identifier le sous-type. Elle permet aussi une meilleure sensibilité du résultat.
PCR asymétrique
C'est l'amplification par PCR en présence d'une faible quantité d'une des amorces. Elle permet le séquençage direct des fragments amplifiés. Pendant les 20 à 25 premiers cycles, l'ADN double brin est généré, jusqu'à épuisement de l'amorce limitante et de l'ADN simple brin est produit pendant les 5 à 10 derniers cycles. Les rapports d'amorces utilisés sont de 50 pmole/1-5 pmoles/100 µL (1-3 pmoles simple brin après 30 cycles).
PCR à asymétrique thermique entrelacée
La PCR à asymétrique thermique entrelacée (TAIL-PCR ou Thermal asymmetric interlaced PCR) est un protocole complexe alliant les principes de la PCR emboîtée, de la PCR asymétrique par le Tm des amorces, la succession de plusieurs types de cycle favorisant l’hybridation de telle ou telle amorce, et des amorces dégénérées. Le but est d’obtenir un amplicon final spécifique d’une séquence d’ADN dont seule une extrémité est connue généralement dans l’optique de séquencer la partie inconnue. Cette méthode est très utilisée pour la marche sur chromosome ou la caractérisation des séquences variables des immunoglobulines.
PCR en gradient de température
Il s’agit d’une technique aidant à la mise au point d’un nouveau protocole de PCR. Elle nécessite des thermocycleurs capables d’assurer des températures différentes, pour une même étape, aux différents échantillons. Elle est surtout utilisée pour optimiser l’étape d’hybridation, notamment en PCR multiplexe.
Les PCR quantitative
PCR semi-quantitative
PCR en temps réel ou PCR quantitative
La PCR en temps réel (Real-time PCR) est une révolution dans l’utilisation de la PCR, cette technique consiste à mesurer la quantité d’ADN polymérisé à chaque cycle (temps réel) grâce à un marqueur fluorescent. Elle permet par son principe de faire des mesures quantitatives (expliquant l'appellation PCR quantitative, qPCR) mais elle nécessite des thermocycleurs particuliers. Il ne faut surtout pas la confondre avec la RT-PCR (Reverse Transcription PCR), on préférera donc les appellations PCR quantitative ou qPCR. Certaines expériences en PCR compétitive ou PCR radioactive permettent l'obtention de mesure quantitative exploitable.[réf. souhaitée]
PCR en point final
La PCR en point final (end point PCR) est un terme qui est apparu en opposition à la PCR en temps réel. Il désigne toutes les tentatives de quantification à partir du produit final d’une réaction de PCR.
PCR par essais
La PCR par essais (Touchdown PCR) est un protocole utilisé pour amplifier de l'ADN faiblement représenté et/ou subissant une compétition sur leurs amorces par des produits de pseudo-gène. Il consiste à avoir une température d’hybridation très haute lors des premiers cycles afin d’assurer une forte stringence et donc une amplification spécifique. Une fois que la séquence d'intérêt devient majoritaire vis-à-vis de ses compétiteurs, la température d’hybridation est progressivement abaissée afin d’assurer une meilleure efficacité de PCR. L’efficacité n’étant pas constante tout au long de la réaction, il est très difficile (impossible ?) de pouvoir obtenir un résultat quantitatif sans biais.
PCR sur colonie
La PCR sur colonie (Colony PCR) est un protocole qui permet d'amplifier de façon simple de l'ADN de micro-organisme (bactéries, archées ou levures) en inoculant directement la colonie dans le milieu réactionnel de la PCR. Durant les premières étapes de dénaturation, les cellules sont lysées et leur ADN est libéré dans le milieu réactionnel. L'ADN ainsi libéré peut alors servir de matrice.
La PCR sur colonie est utilisée notamment pour vérifier l'efficacité d'une transgénèse. Elle fut très utilisée lors de la construction des BAC dans les grands programmes de séquençage.
La PCR sur colonie est largement utilisée en recherche microbiologique afin de caractériser sur le plan phylogénétique les souches étudiées.
Après transcription inverse
RT-PCR
La RT-PCR (Reverse Transcriptase PCR) est une technique qui associe une transcription inverse (RT) suivie d’une PCR. Elle permet de synthétiser le brin complémentaire d’un ARN avec des désoxyribonucléotides en utilisant une ADN polymérase ARN dépendante (transcriptase inverse). Cet ADNc est généralement destiné à être amplifié par PCR (l'ADNc étant plus stable, il permet plus de liberté que les ARN pour les analyses suivantes).
RT-PCR en une étape
La RT-PCR en une étape (single step RT-PCR) est un protocole mélangeant les réactifs de RT et de PCR afin que les deux étapes puissent se faire sans avoir à ouvrir le tube. Cela permet de réduire le risque de contamination ou d’inversion d’échantillon mais il est plus difficile d’optimiser le milieu réactionnel pour chaque étape. Cela induit en outre un risque de biais pour normaliser l’étape de RT car cela implique d'utiliser la PCR multiplexe.
RT-PCR in situ
La RT-PCR in situ est une méthodologie consiste à réaliser la RT-PCR non pas sur des molécules en solution mais sur des coupes histologiques. Bien que les résultats ne soient que semi-quantitatifs, ils apportent en outre une information sur la localisation des transcrits dans le tissu.
RT-PCR quantitative
La RT-PCR quantitative (qRT-PCR) est une technique destinée à pouvoir quantifier un type d’ARN initialement présent dans un échantillon. Ce terme désigne l'utilisation de deux techniques successivement, une Transcription inverse suivie d’une PCR en temps réel. Objectif principal de la plupart des biologistes, elle soulève de vives polémiques quant à l’utilisation de calibrateur externe ou interne (gène de ménage). À l’exception de complexes protocoles utilisant des calibrateurs externes homologues compétitifs, elle ne permet qu’une quantification relative.
RT-PCR sur une cellule
La RT-PCR sur une cellule (single-cell RT-PCR) est une méthodologie qui permet d’étudier les transcrits d’une cellule unique, obtenue par patch-clamp, microdissection laser ou triage de cellules par activité de fluorescence (FACS en anglais pour Fluorescence activated cell sorting). Cette précision peut être nécessaire lorsque le tissu n’est pas homogène (cellules tumorales, feuillets cellulaires bien différenciés, etc.) mais la quantification va devenir moins précise à cause d’une amplification des effets stochastiques et nécessite donc une multiplication des mesures.
PAN-AC
Une autre technique d'amplification isotherme : "Polymérisation des Acides Nucléiques pour Apoptose Controlée" . Les auteurs prétendent que cette technique pourrait être mise en oeuvre dans des cellules vivantes, et ainsi soigner diverses pathologies (HIV, Malaria, cancers...)[1] mais ces affirmations n'ont été corroborées par aucune autre équipe pour le moment.
TP-PCR
La TP-PCR (TP pour Triplet Repeat primed), une variante complexe de la PCR, a été mise au point par Jon Warner en 1996 et est utilisée dans les amplifications des gènes comportant des triplets répétés, comme dans le cas de la Dystrophie myotonique de Steinert. La combinaison PCR-Séquençage ne peut pas être utilisée dans ces cas car la Taq polymérase fait des erreurs de réplication.
Amplification hélicase-dépendante
L'amplification hélicase-dépendante (HDA ou helicase-dependent amplification) est une technique récente proche de la PCR, où la déshybridation induite par la température est assurée par une hélicase.
Autres techniques
- ARDRA
- Hybridation in situ
- LAMP
- Microarray
- Nasba
- Northern blot
- RACE-PCR
- RNAse Protection Assay (RPA)
- Southern blot
- TMD
- ADN branché
- LCR
- PAN-AC (voir : http://www.lab-rech-associatives.com/pages/agi.php )
Sources
- ↑ (en) F. David et E. Turlotte, « An Isothermal Amplification Method », dans Comptes-Rendus de l'Académie des Sciences, vol. 321, 1998, p. 909-914
Bibliographie
- (fr) Elyse Poitras et Alain Houde. La PCR en temps réel: principes et applications. Reviews in Biology and Biotechnology (Canada). Vol.2, No 2, December 2002. pp.2-11
- Articles anglophones
- (en) Baltimore D. RNA-dependent DNA polymerase in virions of RNA tumor viruses. 1970. Nature 226, 1209–1211
- (en) Birch DE. Simplified hot start PCR. 1996. Nature;381:445–6.
- (en) Friedberg EC. The eureka enzyme: the discovery of DNA polymerase. Nat Rev Mol Cell Biol. 2006 Feb;7(2):143-7.
- (en) Higuchi R, Dollinger G, Walsh PS, Griffith R. Simultaneous amplification and detection of specific DNA sequences. 1992. Biotechnology;10:413–7.
- (en) Detection of specific polymerase chain reaction product by utilizing the 5’–3’ exonuclease activity of Thermus aquaticus DNA polymerase Holland PM, Abramson RD, Watson R, Gelfand DH. Proc Natl Acad Sci USA 1991;88:7276–80.
- (en) Kornberg, A., Lehman, I. R., Bessman, M. J & Simms, E. S. Enzymatic synthesis of desoxyribonucleic acid. 1956. Biochim. Biophys. Acta 21, 197–198
- (en) Lay MJ, Wittwer CT. Real-time fluorescence genotyping of factor V Leiden during rapid-cycle PCR. 1997. Clin Chem;43:2262–7.
- (en) Liu YG, Whittier RF. Thermal asymmetric interlaced PCR: automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking. 1995. Genomics. Feb 10;25(3):674-81.
- (en) Mullis K, Faloona F, Scharf S, Saiki R, Horn G, Erlich H. Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction. Cold Spring Harb Symp Quant Biol. 1986;51 Pt 1:263-73.
- (en)Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, Erlich HA. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 1988 Jan 29;239(4839):487-91.
- (en) Stoflet ES, Koeberl DD, Sarkar G, Sommer SS. Genomic amplification with transcript sequencing. Science. 1988 Jan 29;239(4839):491-4.
- (en) Temin HM & Mizutani S. RNA-dependent DNA polymerase in virions of Rous sarcoma virus. 1970. Nature 226, 1211–1213 (1970).
- (en) Watson JD & Crick FC. A structure for deoxyribose nucleic acid. Nature. 1953 171, 737–738
- (en) Warner JP et al. A general method for the detection of large CAG repeat expansions - Journal of Medical Genetics, December 1996, Vol. 33, No 12, pp. 1022 – 1026
- (en) Wittwer CT, Herrmann MG, Moss AA, Rasmussen RP. Continuous fluorescence monitoring of rapid cycle DNA amplification. 1997. Biotechniques;22:130–1, 134–8.
Voir aussi
Articles connexes
Liens externes
- Animation
- Animation PCR : principe de la PCR et de la PCR en temps réel et comparaison de ces techniques.
- Animation (université de Lyon)
- Animation PCR - (Flash)
- Portail de la biologie cellulaire et moléculaire
- Portail de la biochimie
- Portail des technologies
Catégories : Réaction en chaîne par polymérase | Biologie moléculaire | Technique de laboratoire
Wikimedia Foundation. 2010.