- Homologie (biologie)
-
Homologie (évolution)
 Pour les articles homonymes, voir Homologie.
Pour les articles homonymes, voir Homologie.En biologie de l'évolution, une homologie désigne un lien évolutif entre deux traits (en général, anatomiques) observés chez deux espèces différentes, qui est due au fait que toutes deux l'ont hérité d'un ancêtre commun. Ces traits sont alors dits homologues. Ce peut être des caractères anatomiques, ou moléculaires (protéines homologues). Ce terme s'étend aussi aux séquences génétiques ( de l'ADN ).
Sommaire
Niveau anatomique
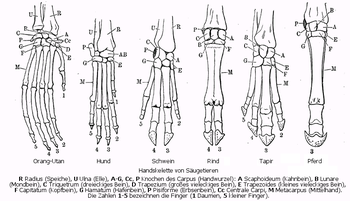 Le membre des vertébrés est une homologie, car on observe le même plan d'organisation de la "main" ou partie terminale du membre antérieur, respectivement chez :
Le membre des vertébrés est une homologie, car on observe le même plan d'organisation de la "main" ou partie terminale du membre antérieur, respectivement chez :
l’orang-outan, le chien, le cochon, les bovidés, le tapir et le cheval.C'est le cas, par exemple, de la patte antérieure d'un cheval et de la nageoire pectorale d'un dauphin, qui ont comme ancêtre commun un mammifère (qui, il y a 80 millions d'années, ressemblait probablement à un chevrotain[1]), qui possédait comme ces deux animaux quatre membres et c'est le membre antérieur de ce mammifère primitif qui a donné la patte du cheval et la nageoire du dauphin.
Lorsque deux traits apparaissent indépendamment dans deux taxons sans être l'héritage d'un ancêtre commun comme les ailes des insectes et celles des oiseaux, on parle d'analogie.
Niveau moléculaire
Des protéines homologues sont des protéines dont les gènes qui les codent ont une origine commune. On reconnaît 2 protéines homologues car elles ont des structures spatiales proches et des séquences en acides aminés qui présentent des similarités. Les fonctions de ces protéines peuvent être plus ou moins semblables.
On peut trouver des protéines homologues dans des espèces différentes, c'est le cas des globines des vertébrés. Cela traduit l'origine commune de plusieurs espèces et donc l'évolution. On peut trouver des protéines homologues dans une même espèce, c'est le cas des globines alpha et bêta qui composent l'hémoglobine humaine. Cela démontre que l'évolution peut être due à une complexification des génomes.
Orthologue
Deux séquences homologues de deux espèces différentes sont orthologues si elles descendent d'une séquence unique présent dans le dernier ancêtre commun aux deux espèces. Le terme orthologue a été proposé par Walter Fitch en 1970.
Paralogue
Deux séquences homologues au sein d'une espèce sont paralogues si elles résultent d'une duplication génique.
Inparalogue
Deux séquences paralogues au sein d´une espèce sont inparalogues si l´évènement de duplication a eu lieu après la spéciation[2].
Outparalogue
Deux séquences paralogues au sein d´une espèce sont outparalogues si l´évènement de duplication a eu lieu avant la spéciation.
Ohnologue
Deux séquences paralogues sont ohnologues si elles résultent d´un évènement de duplication complète du génome. Le terme ohnologue a été proposé par Ken Wolfe en hommage à Susumu Ohno[3].
Xénologue
Deux séquences homologues sont xénologues lorsqu´elles résultent d´un transfert horizontal de gène entre deux organismes.
Références
- ↑ (en) Placental mammal diversification and the Cretaceous-Tertiary boundary. Springer MS, Murphy WJ, Eizirik E, O'Brien SJ. Proc Natl Acad Sci U S A. 2003 Feb 4;100(3):1056-61.
- ↑ Sonnhammer EL, Koonin EV. Orthology, paralogy and proposed classification for paralog subtypes. Trends Genet. 2002 Dec;18(12):619-20 [1]
- ↑ Ken Wolfe (2000) Robustness—it's not where you think it is. Nature Genetics May;25(1):3-4. [2]
Liens internes
- analogie
- Anatomie comparée
- Caractère dérivé et caractère ancestral
- Phylogénétique
- Synapomorphie
- Homoplasie
- Portail de la biologie
Catégorie : Théorie sur l'évolution
Wikimedia Foundation. 2010.

