- Centromériques
-
Centromère
 Chromosome.
Chromosome.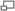
(1) Chromatide. Chacune formée d'un brin d'ADN parental et d'un brin d'ADN néoformé obtenu après réplication durant la phase S ; (2) Centromère. Le point de contact des deux chromatides, et le point de séparation lors de la mitose ; (3) Bras court ; (4) Bras longLe centromère est le site de constriction du chromosome dont les fonctions sont essentielles à la vie des cellules eucaryotes, en particulier (1) relier les deux chromatides soeurs entre elles, entre la réplication et l'anaphase, (2) d'assurer l'assemblage protéique du kinétochore à son niveau, lui même responsable de (3) la ségrégation équitable des chromatides sœurs entre les deux cellules filles lors de la division cellulaire.
Les centromères de la plupart des organismes eucaryotes sont composés d'ADN répété et portent des marques épigénétiques particulières. En période interphasique du cycle cellulaire, les centromères sont localisables à proximité du nucléole.La composition en ADN
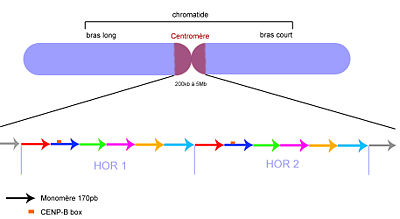
La séquence d'ADN répétée des centromères varie énormément d'une espèce à l'autre. Généralement l'ADN y est répété en tandem sur plusieurs kilobases. Chez l'Homme, l'ADN répété est principalement de l'ADN alpha-satellite, dont le motif unitaire (monomère) fait 171 nucléotides de long. Ces monomères sont répétés un très grand nombre de fois. C'est pour cela qu'on les nomme "higher Order repeat" (HOR). Ces HOR sont alignés de manière ininterrompue dans la même orientation sur des distances très importantes, allant de 40kb à 5Mb. Ces HOR sont très homogènes au niveau de leur séquence: ils partagent 99.8% d'identité de séquence, alors que les monomères au sein de ces HOR ne partagent qu'entre 60 et 80% d'identité de séquence. Cette organisation est présentée figure 2. Outre cette organisation en ADN répété très homogène et ininterrompue, des séquences particulières semblent être primordiales pour l'assemblage du complexe du kinétochore, qui donne au centromère sa fonction essentielle lors de la division cellulaire: la capacité à ségréger équitablement les chromatides dans les cellules filles. C'est le cas de la "boite CENP-B" (CENP-B box), longue de 17pb, capable de lier la protéine CENP-B, nécessaire à l'assemblage de ce complexe protéique. La présence de ces séquences d'attache particulières, présentent dans chaque HOR, ainsi que l'organisation en ADN hautement répété était des caractéristiques des centromères humains, et de bon nombre d'autres espèces eucaryotes. Cependant, des observations récentes sur la formation de néo-centromères en absence d'ADN alpha-satellite incitent à penser que la séquence d'ADN au centromère n'est pas la seule responsable de sa fonction[1].
Les marques épigénétiques
Les marques épigénétiques sont l'ensemble des modifications réversibles et héritables qui peuvent avoir lieu sur la chromatine. Ces marques peuvent être des modifications chimiques des Histones (méthylation, acétylation, phosphorylation ...) ou de l'ADN, ou encore des variants d'histones. C'est le cas au niveau du centromère, puisque l'Histone H3 est remplacé par un de ses variants, la protéine CENP-A. Cette protéine CENP-A est retrouvée dans tous les centromères humains, ainsi que dans les néo-centromères. Ce variant d'histone, ainsi que de nombreuses autres modifications épigénétiques, donnent une structure particulière à la chromatine centromérique. Elle semble plus lâche que l'hétérochromatine (qui interdit la transcription en raison de son trop haut degré de compaction) et plus compacte que l'euchromatine.
Notes et références
- ↑ Schueler M.G and Sullivan B.A. "Structural and functional dynamics of Human Centromeric Chromatin" Annu. Rev. Genomics Hum. Genet. 2006. 7:301-13 at genom.annualreviews.org
- Portail de la biologie cellulaire et moléculaire
Catégories : Génétique | Chromosome
Wikimedia Foundation. 2010.