- Q-PCR
-
PCR quantitative
La PCR quantitative (synonymes : PCR en temps réel, QPCR) permet de mesurer la quantité initiale d'ADN. En réalité, la PCR quantitative mesure le nombre d'amplicon(portion d'ADN définie par un couple d'amorces).
Il existe diverses techniques permettant de quantifier l'ADN en biologie moléculaire. On peut utiliser la spectromètrie à 260 nm. Une autre technique fiable permettant la quantification de l'expression d'un gène est le Northern blot. Cette technique a été développée historiquement en utilisant l'hyridation de sondes radioactives ou fluorescente sur l'ADN amplifié. Le Northern blot nécessite des sondes radioactives ce qui pose des problèmes au niveau de la sécurité (radioactivité) et du temps d'exposition des films radiographiques (long).Sommaire
Historique
En PCR en point final
Beaucoup de personnes s'affranchissent de mettre en œuvre les techniques du Northern blot et quantifient à l'aide de la technique de la PCR en point final. Cette quantification est faite selon le postulat que plus il y avait d'ADN au départ, plus il y en a à l'arrivée, à la fin des 40 cycles d'amplification. On quantifie ensuite grossièrement à la lecture des gels d'agarose, grâce à la comparaison des intensités des bandes obtenues avec celles d'un marqueur de poids moléculaire. L'œil humain ne permettant pas de discriminer de façon absolue les différences d'intensité entre les bandes, on peut avoir recours à un logiciel de d'analyse d'images, tel ImageJ. Cependant, la précision gagnée dans la lecture de l'intensité de la bande peut-être compensée par une perte de précision due au côté arbitraire du placement de la ligne de base et à la présence d'un bruit de fond non-homogène sur l'image.
La PCR semi-quantitative
La PCR semi-quantitative est basée sur l'interruption de la PCR en plusieurs cycles qui correspondent à la phase stationnaire (la quantité d'ADN augmente très doucement car il y a peu de d'ADN matrice), la phase exponentielle (croissance rapide) et au plateau (diminution de la quantité de réactifs). Pour un échantillon, il est possible d'estimer la quantité initiale d'ADN si l'on dispose d'un échantillon où la quantité d'ADN est connue (étalon). Leur amplification par PCR, l'arrêt de la réaction entre le 20e et le 30e cycle PCR et la comparaison entre les intensités lumineuses des produits PCR marqués au BET permettent d'estimer la quantité initiale d'ADN. Pour deux échantillons, il est possible de comparer leur quantité d'ADN initiale en arrêtant la PCR avant le plateau de la PCR (<30 cycles).
La PCR compétitive
En PCR en temps réel
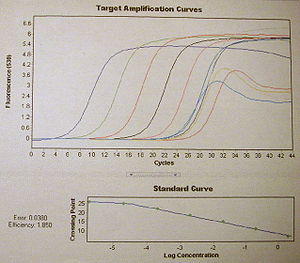
Le principe de la PCR en temps réel repose sur la possibilité de suivre la quantité d'ADN présente dans la réaction à tout instant et non à la fin de la PCR (PCR point final)ou au cycle n<40 (PCR semi-quantitative). Des sondes fluorescentes se fixent soit sur l'ADN double brin (technologie SYBR) ou sur une séquence d'ADN précise (technologie Taqman et Beacon). Ces sondes ne fluorescent qu'une fois fixées à l'ADN (soit à cause d'un "quencher" soit car la fluorescence nécessite un ADN double brin). Un seuil de fluorescence est établie par le programme de la machine de PCR en temps réel. Une fois que la quantité d'ADN permet aux sondes fluorescentes de dépasser ce seuil alors on obtient un numéro de cycle PCR appelé "Ct"pour "Cycle Threshold" ou cycle seuil. C'est cette valeur qui est à la base des calculs pour quantifier l'ADN de façon absolue ou relative. Il est important de connaître l'efficacité E de la PCR. Pour cela, on effectue une PCR en temps réel sur des échantillons de dilution croissante pour obtenir une courbe étalon correspondant au couple d'amorces utilisé (spécifiques du locus d'intérêt). Par exemple, une série de dilution au 1/2 (D<inf>n+1<inf>=D<inf>n<inf>/2) doit, en théorie, donner des courbes d'amplification décalées d'un cycle PCR à chaque fois. Si tel est le cas, la réaction a alors une efficacité égale à 2 (la quantité d'ADN double à chaque cycle). En pratique, le programme de la machine de PCR en temps réel peut calculer l'efficacité E de la réaction. Plus souvent, une PCR en temps réel sur une série de dilution avec une quantité d'ADN initiale connue permet de calculer l'efficacité de la réaction. Les Ct sont placées sur un graphe en échelle logarithmique et l'équation de la régression linéaire passant par ces points donne l'éfficacité (c'est le coefficient directeur).
Quantification absolue
Durant la partie de la courbe en sigmoïde de l'échantillon, après la ligne de seuil de détection, au moment où l'amplification est exponentielle, on a :
Qn = Qo.En (1)
où :
Q est la quantité d'ADN
n est l'indice du nième cycle
0 est l'indice du cycle de départ
E est l'efficacité de la réactiondonc au cycle de seuil (ct ou threshold cycle), premier cycle au dessus de la ligne de base :
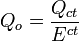
Quantification relative
On établit un rapport R entre la quantité d'ADN de départ d'un échantillon et celle d'un témoin, qui n'a pas subi de traitement. Au seuil de détection, on a :
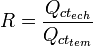
de (1), on déduit donc :
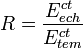
Soit :
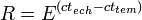 ou : R = EΔct
ou : R = EΔctQuantification relative corrigée par un gène de référence
Le gène de référence est un gène qui n'est pas induit par le traitement que l'on fait subir pour mesurer la variation du gène cible. Effectuer une correction à partir d'un gène de référence permet d'éliminer les effets de fluctuations.
Bibliographie
- (en) Guide pratique de QPCR de l'Université de Copenhague
- (en) Tutoriels et Informations Techniques sur la PCR quantitative
Catégorie : Réaction en chaîne par polymérase
Wikimedia Foundation. 2010.